21. Évolution de la diète
Introduction
Dans ce chapitre, nous aborderons l’évolution du système digestif chez les humains et d’autres mammifères. Nous verrons comment deux enzymes qui permettent de digérer des aliments ont évolué dans différentes populations en association avec leurs ressources alimentaires.
Objectifs d’apprentissage
À la fin de ce chapitre, vous serez en mesure de :
- Comprendre le rôle des enzymes impliquées dans la digestion
- Définir ce que sont des variants structuraux génomiques
- Expliquer l’effet potentiel du nombre de copies du gène codant pour l’amylase sur la quantité d’amylase produite dans la salive.
- Expliquer le cas du gène AMY1 dont le nombre de copies varie entre les populations d’humains selon leur diète
- Expliquer comment les données génomiques suggèrent une évolution indépendante du nombre de copies du gène AMY entre des espèces de mammifères domestiqués
- Expliquer le lien entre la persistance de la lactase et la tolérance au lactose à l’âge adulte chez l’humain
- Décrire l’étendue de la variation interindividuelle dans la persistance de la lactase à l’âge adulte chez les humains et son origine
- Savoir interpréter un diagramme représentant un pedigree afin de déterminer comment un trait physiologique est transmis entre les générations
- Expliquer l’effet potentiel d’une mutation dans la séquence codante du gène codant pour la lactase
- Expliquer l’effet potentiel d’une mutation dans la séquence régulatrice du gène codant pour la lactase
- Appliquer une analyse fonctionnelle au cas de la mutation de la séquence régulatrice de la lactase
- Expliquer les observations qui ont mené à l’hypothèse d’une évolution convergente de la persistance de la lactase chez différentes populations humaines
- Expliquer les causes proposées de l’évolution de la persistance de la lactase
Notions clés
- La digestion
- Les enzymes impliquées dans la digestion des glucides
- Évolution d’enzymes impliquées dans la digestion des glucides
- L’amylase
- L’amylase salivaire
- Les enzymes du tube digestif
- Les variants structuraux génomiques chez l’humain
- Variation du nombre de copies du gène codant pour l’amylase
- Association de la variation du nombre de copies du gène codant pour l’amylase et de la diète chez l’humain
- Évolution indépendante du nombre de copies de l’amylase pancréatique chez les chiens
- Évolution indépendante de la duplication des gènes codant pour l’amylase et du nombre de copies chez les mammifères domestiqués
- Association entre la diète, le nombre de copies de gènes codant pour l’amylase et l’amylase salivaire
- La lactase
- La persistance de la lactase à l’âge adulte chez les humains
- Les populations humaines ont différents niveaux de persistance de la lactase
- Adaptation au lactose dans la diète à l’âge adulte chez l’humain
- Utilisation de pedigree humain pour déterminer le mode d’hérédité
- Déterminer si un trait est dominant ou récessif à l’aide d’un pedigree
- Mutation dans la séquence codante du gène codant pour la lactase
- Mutation dans la séquence régulatrice du gène codant pour la lactase
- Quantification de l’effet fonctionnel de la mutation
- Évolution convergente de la persistance de la lactase
- Lien entre l’évolution de la persistance de la lactase et les développements socioculturels chez l’humain.
1. La digestion
La section 1 et la sous-section 1.1 sont adaptées de [1].
Le processus de digestion mécanique est relativement simple. Il implique la dégradation physique des aliments mais ne modifie pas leur composition chimique. La digestion enzymatique, quant à elle, est un processus complexe qui réduit les aliments en ses éléments constitutifs chimiques, qui sont ensuite absorbés pour nourrir les cellules du corps.
Les grosses molécules alimentaires (par exemple, les protéines, les lipides, les acides nucléiques et les amidons) doivent être décomposées en sous-unités suffisamment petites pour être absorbées par le tube digestif. Chez l’humain, cela est accompli par des enzymes par hydrolyse, résumées dans la figure 21.1.
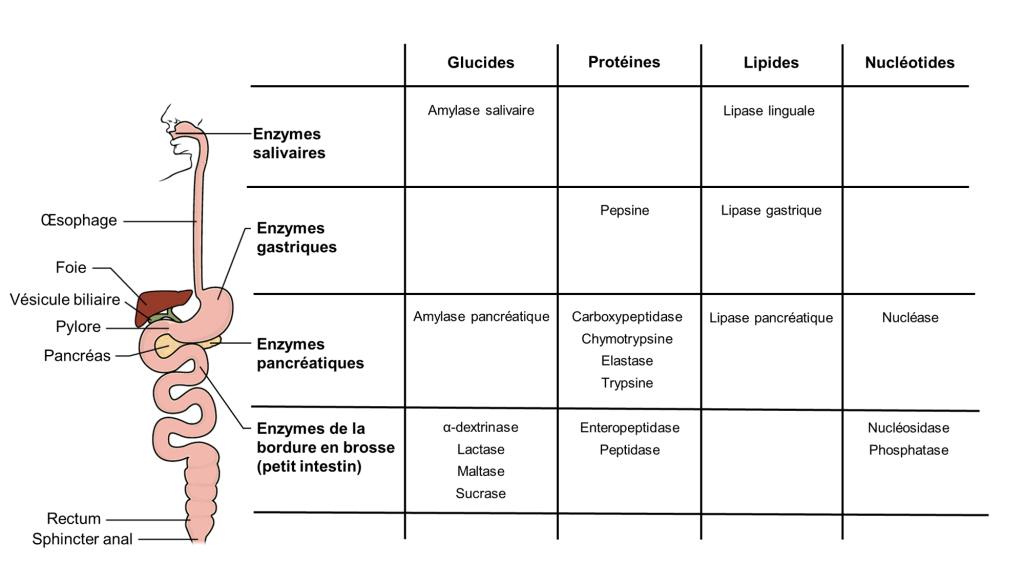
1.1 Les enzymes impliquées dans la digestion des glucides
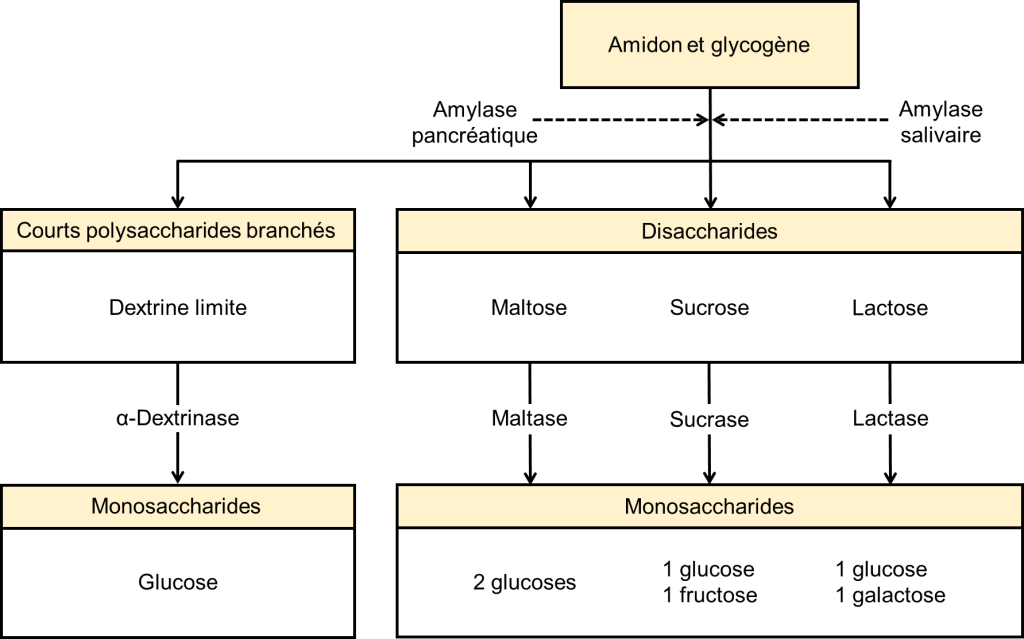
Le régime d’une personne américaine moyenne est composé d’environ 50 % de glucides, qui peuvent être classés en fonction du nombre de monomères de sucres simples qu’ils contiennent : d’un côté les monosaccharides et disaccharides, et d’un autre les polysaccharides, qui sont des chaines de monosaccharides. Le glucose, le galactose et le fructose sont les trois monosaccharides couramment consommés et facilement absorbés. Le système digestif des humains est également capable de décomposer les disaccharides suivants : le saccharose (sucre de table ordinaire : glucose + fructose), le lactose (sucre du lait : glucose + galactose) et le maltose (sucre de grain : glucose + glucose). Il en va de même pour les polysaccharides, le glycogène et l’amidon (figure 21.2). Cependant, chez les humains, le corps ne produit pas d’enzymes capables de décomposer la plupart des polysaccharides fibreux, comme la cellulose. Bien que les polysaccharides non digestibles n’apportent aucune valeur nutritive, ils fournissent des fibres alimentaires, qui aident au mouvement des aliments dans le tube digestif.
1.2 Évolution d’enzymes impliquées dans la digestion des glucides
Comme tous les autres systèmes physiologiques, on observe une fascinante diversification entre les espèces de leur système digestif, selon les ressources alimentaires qu’elles utilisent. Que l’on pense aux variations morphologiques du système digestif entre les omnivores et les carnivores ou aux modifications liées à l’herbivorie chez les ruminants, la diversité des adaptations physiologiques est manifeste. Il existe aussi des variations entre les individus d’une même espèce dans la physiologie digestive. Ces variations semblent être associées aux ressources alimentaires utilisées et pourraient être des adaptations. Nous nous concentrerons maintenant sur deux enzymes impliquées dans la digestion des glucides et leur évolution chez les mammifères en lien avec la diète : l’amylase et la lactase.
2. L’amylase
2.1 L’amylase salivaire
La sous-section 2.1 est adaptée de [4].
La salive est essentiellement composée d’eau (95,5 %). Les 4,5 % restants sont un mélange complexe d’ions, de glycoprotéines, d’enzymes, de facteurs de croissance et de mucus. L’ingrédient le plus important de la salive du point de vue de la digestion chez l’humain est l’enzyme amylase salivaire, qui initie la dégradation des glucides à longue chaine qui forment l’amidon (figure 21.2). Les aliments ne passent pas assez de temps dans la bouche pour permettre à tout l’amidon de se décomposer, mais l’amylase salivaire continue d’agir jusqu’à ce qu’elle soit inactivée par les acides gastriques.
2.2 Les enzymes du tube digestif
La sous-section 2.2 est adaptée de [1].
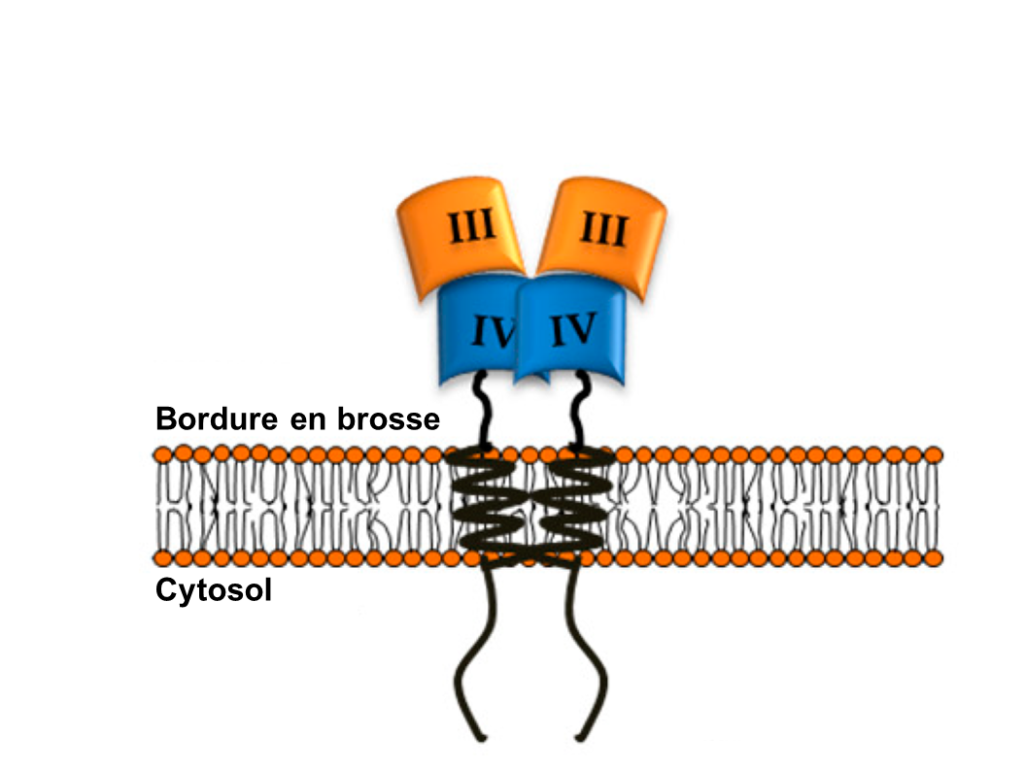
L’amylase pancréatique agit dans l’intestin grêle où elle hydrolyse l’amidon (figure 21.2). Après que les amylases aient décomposé l’amidon en fragments plus petits, l’enzyme α-dextrinase de la bordure en brosse commence à agir sur l’α-dextrine, cassant une unité de glucose à la fois (figure 21.3). Trois enzymes de bordure en brosse hydrolysent le saccharose, le lactose et le maltose en monosaccharides. La sucrase divise le saccharose en une molécule de fructose et une molécule de glucose; la maltase décompose le maltose et le maltotriose en deux et trois molécules de glucose, respectivement; et la lactase décompose le lactose en une molécule de glucose et une molécule de galactose (figure 21.2).
Exercices
Digestion enzymatique
2.3. Les variants structuraux génomiques
Afin de discuter de l’évolution de l’activité de l’amylase, nous devons d’abord revenir sur un concept de génétique, soit les variants structuraux génomiques. Ceux-ci sont une forme de diversité génétique. Bien que la variation génomique moyenne entre deux humains soit de 0,1 % en termes de variants mononucléotidiques (SNPs), en prenant en compte les variants structuraux, la variation interindividuelle passe à 1,5 %! C’est donc une source incroyable de diversité à l’intérieur de notre espèce. On savait déjà depuis la fin des années 50 qu’il existe des variants structuraux chez les humains ainsi que chez la drosophile depuis les années 70. Au milieu des années 2000, les biologistes ont obtenu assez de données sur plusieurs génomes d’humain, pour réaliser qu’en fait, les variations du nombre de copie sont très courantes entre les individus et ce à l’échelle du génome. Ces différences émergent de la duplication de segments du génome mais aussi de la délétion de certaines portions. On a le plus d’information pour les humains parce qu’on a mis des efforts et du financement pour séquencer plusieurs génomes humains, mais c’est vrai pour la plupart des organismes. Avec les données actuelles, nous savons qu’au moins 1000 gènes présentent une variation du nombre de copies (CNV pour Copy Number Variation) chez l’humain, c’est-à-dire que pour ces 1000 gènes, certaines personnes ont plus ou moins que 2 copies [6-8].
Une variation du nombre de copies d’une région de génome est souvent associée à un changement de l’abondance des protéines codées par les gènes qui s’y trouvent (mais pas toujours). Le changement de dosage lié à un changement de nombre de copies peut avoir des effets mineurs ou nuls selon la fonction de la protéine affectée. Cependant, certaines maladies et certains syndromes chez l’humain sont associés à une augmentation du nombre de copies d’un gène, entre autres lorsqu’une troisième copie d’un certain chromosome est présente [6]. Dans ce cas, le changement de dosage a des effets majeurs négatifs. En fait, beaucoup de cas ne sont pas répertoriés, car ils sont non viables aux premiers stades de développement. Cependant, le changement de dosage a parfois un effet bénéfique. En effet, dans certains cas, avoir plus de protéine implique que l’activité totale de cette protéine est augmentée et cela peut avoir une incidence positive sur la performance et le fitness. Nous allons voir un exemple où un plus grand nombre de copies produit un plus grand nombre de protéines, ce qui change le phénotype de l’organisme d’une façon qui pourrait être bénéfique pour son fitness.
Aubin-Horth, N. (2024). Écophysiologie évolutive: les variants structuraux génétiques. [Vidéo]. Youtube. CC-BY. https://youtu.be/4f_9OMlkSDY?si=7H4szymiDcsR5R_1
Vous trouverez la transcription de l’audio de cette capsule en cliquant sur ce lien: Transcription_chapitre21
2.4 Variation du nombre de copies du gène codant pour l’amylase
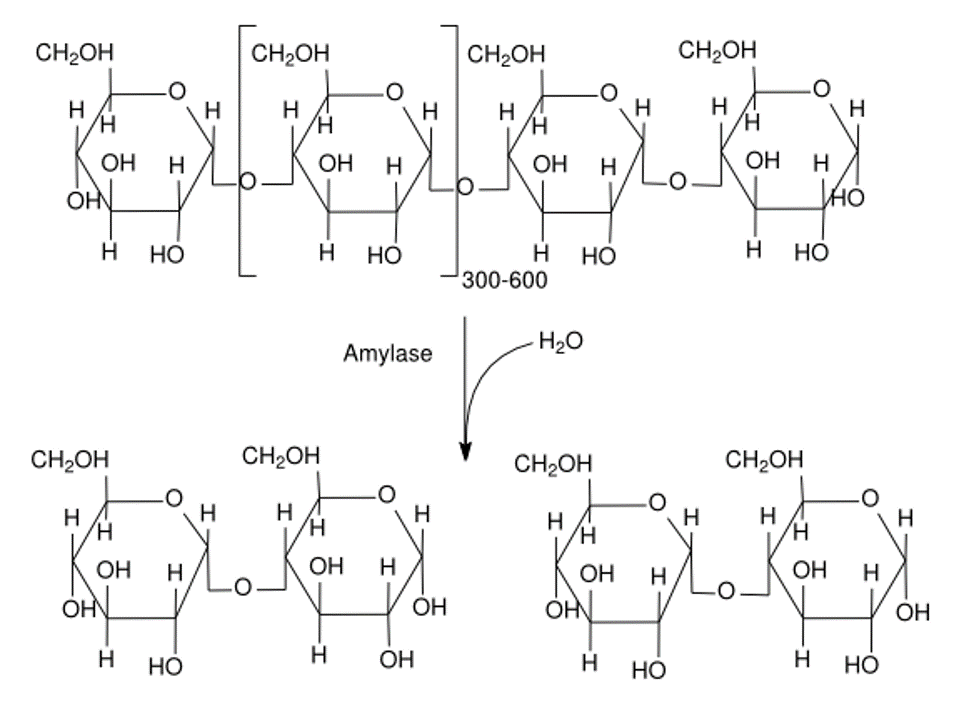
L’amidon est un polysaccharide provenant des plantes. Il est composé de glucose et est donc une source potentielle d’énergie pour les animaux qui ingèrent des aliments riches en amidon. Cependant, il forme une structure semi-cristalline insoluble et par conséquent il ne peut pas être dissous dans l’eau ou dans l’acide gastrique. L’enzyme amylase est nécessaire pour sa digestion, en le brisant en glucides simples (figure 21.4).
Toutes les espèces de vertébrés ont des gènes qui codent pour l’amylase. Une version du gène (AMY2) est exprimée dans le pancréas. L’amylase produite dans le pancréas est sécrétée dans l’intestin grêle où elle peut agir sur l’amidon. Comme nous avons vu chez l’humain, certaines espèces produisent également de l’amylase dans leur salive. L’amylase salivaire peut commencer à décomposer l’amidon dès qu’il est mangé. Cette amylase est le produit du gène AMY1, un paralogue du gène AMY2. Le gène AMY1 a un nouveau patron d’expression dans un nouveau tissu, passant du pancréas aux glandes salivaires. On peut donc proposer que ce soit un cas de néo-fonctionnalisation, c’est-à-dire qu’il y a eu évolution de la séquence régulatrice de cette copie du gène qui fait qu’il est exprimé dans un nouveau tissu.
Ce qui nous intéresse dans cette composante du système digestif, c’est qu’en plus de la duplication du gène lui-même qui a donné AMY1 et AMY2 qui sont exprimés dans des tissus différents, il y a aussi de la variation du nombre de copies entre les individus de la même espèce pour le gène AMY1 salivaire. Il a été démontré dans les années 1980 que le nombre de copies du gène codant pour l’amylase salivaire varie grandement d’un humain à l’autre. Comme vous le savez, les humains sont diploïdes et ils ont donc deux copies de tous leurs gènes (un de chaque parent), sauf pour les gènes sur les chromosomes sexuels. Nous nous attendons donc à voir deux copies d’un gène chez un humain, mais nous avons entre 2 et 15 copies du gène codant pour l’amylase salivaire (figure 21.5). Ces variations du nombre de copies sont le résultat de la duplication de gènes, mais seulement certains individus d’une espèce ont ces duplications dans leur génome. Cette variation du nombre de copies du gène codant pour l’amylase est associée à une variation de la quantité d’amylase dans la salive (figure 21.5). Plus une personne a de copies du gène AMY1, plus elle exprime la protéine amylase dans sa salive. Ils ont cependant aussi noté que le nombre de copies n’expliquait pas toute la variation de la quantité de protéines et ont proposé que le niveau d’expression du gène pourrait aussi avoir évolué différemment entre les individus, par exemple dû à une mutation dans le promoteur du gène. Les biologistes qui ont étudié ces échantillons ont aussi démontré que l’activité de l’amylase salivaire par ml augmente à mesure que le nombre de copies de gènes augmente [10-12].
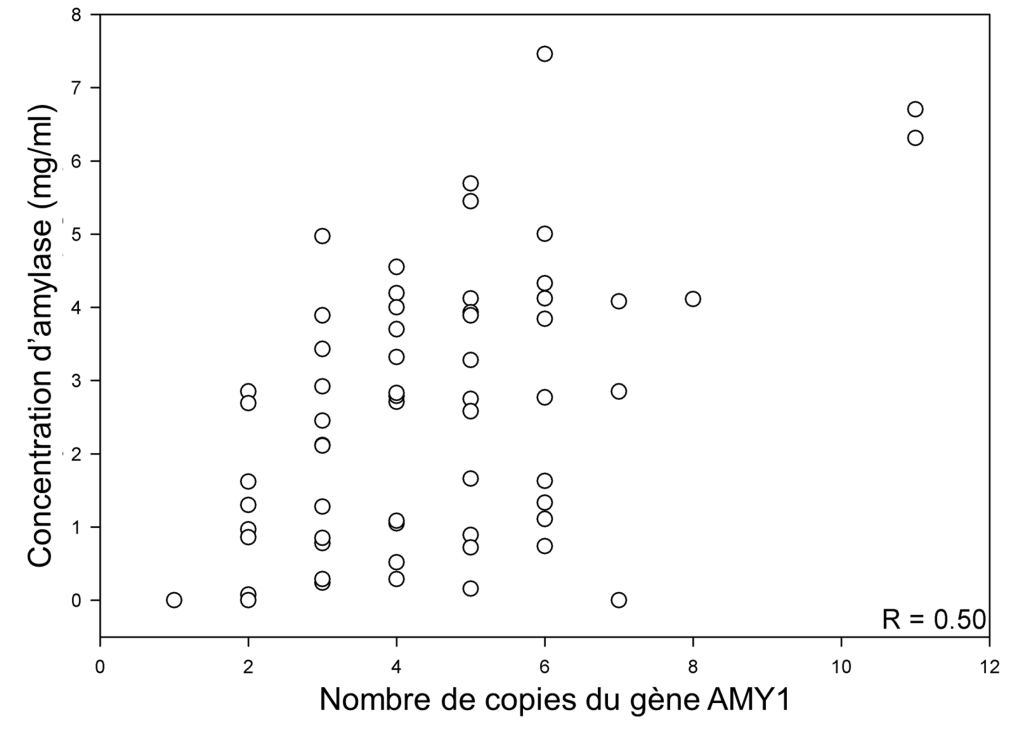
2.5 Association de la variation du nombre de copies du gène codant pour l’amylase et de la diète chez l’humain
Il est possible que ces différences du nombre de copies soient une adaptation à la composition de la diète et à l’utilisation de l’amidon par certaines populations humaines. Les biologistes qui ont émis cette hypothèse se sont basés sur le fait que les variations ne se répartissent pas uniquement selon la géographie. Si on observe un patron selon lequel les populations qui ont le plus grand nombre de copies vivent dans les mêmes régions, cela suggèrerait un patron d’évolution neutre. Si les populations qui ont le plus grand nombre de copies ont la même diète mais ne sont pas géographiquement proches, cela suggèrerait plutôt un patron qui est le résultat de la sélection naturelle. Les biologistes ont montré que des individus vivant en Afrique avaient le même nombre de copies que des individus vivant en Asie. La même chose était vraie pour les populations avec un bas nombre de copies puisqu’on en retrouvait autant en Afrique qu’en Asie. Ils ont de plus découvert une association entre la diète et les variants structuraux génomiques chez ces populations. L’apport en amidon par le riz, les pommes de terre et autres aliments riches en amidon est plus élevé dans les populations qui ont un plus grand nombre de copies, ce qui suggère que la sélection naturelle a favorisé ce grand nombre de copies face à ce type de diète [11]. Cependant, il s’agit d’une corrélation et il faut pouvoir relier la variation dans la quantité d’amylase salivaire avec le fitness avant de pouvoir considérer que ce trait est effectivement une adaptation à une diète riche en amidon. L’observation que la plus grande quantité d’amylase salivaire résulte en une plus grande activité de l’enzyme devrait être directement reliée à la performance et au fitness pour conclure sur ce sujet.
Étant donné la possibilité que cette variation du nombre de copies soit une adaptation chez les humains, des biologistes évolutifs se sont demandé quand sont apparues ces duplications qui ont mené à des variations du nombre de copies du gène AMY. En comparant des génomes de diverses populations humaines et celui des Néanderthaliens, ils ont démontré que cette variation génétique est apparue chez les humains et n’était pas présente chez les Néanderthaliens qui avaient seulement deux copies du gène AMY1. Leur analyse démontre aussi que les humains avaient déjà de multiples copies de ce gène lorsqu’ils ont migré d’Afrique vers les autres régions du globe [13].
2.6 Évolution indépendante du nombre de copies du gène codant pour l’amylase pancréatique chez les chiens
Les humains ont domestiqué de nombreuses espèces au fil de leur évolution récente. Des biologistes se sont demandé si les espèces qui sont en contact étroit avec l’humain pourraient avoir le même type de changement dans leur système digestif, soit un plus grand nombre de copies des gènes AMY. Cette hypothèse était basée sur le fait que lorsque les animaux vivent avec les humains, ils mangent souvent les restes de table et sont donc exposés à une nouvelle diète riche en amidon.
La domestication du chien s’est produite il y a plus de 10 000 ans, peut-être jusqu’à il y a 30 000 ans. Cette domestication s’est faite probablement à plusieurs reprises et dans tous les cas, les canidés qui ont été domestiqués ont partagé l’environnement des humains, incluant leur nourriture. Des biologistes ont étudié les différences dans le génome entre les chiens et les loups, à la recherche de signes de sélection dans le génome du chien, afin de découvrir quelles fonctions biologiques ont changé en association avec la domestication. Elles ont découvert 36 régions génomiques qui pourraient avoir été la cible de la sélection liée à la domestication. Parmi les nombreuses fonctions, plusieurs étaient reliées au développement et au fonctionnement du système nerveux, ainsi qu’au système digestif (métabolisme des lipides et digestion de l’amidon). Elles ont démontré que par rapport au loup, les chiens avaient un plus grand nombre de copies du gène AMY2, qui code pour l’amylase sécrétée par le pancréas et qui est active dans l’intestin. Afin de confirmer l’effet de cette augmentation du nombre de copies sur la digestion, elles ont aussi démontré que les gènes étaient effectivement plus exprimés dans le pancréas chez les chiens et que l’activité de l’amylase de chiens était plus élevée que celle de loups [14].
Cette découverte initiale a amené d’autres biologistes à se demander si de la variation pouvait être présente entre les races de chiens. En effet, les chiens ne sont pas nourris de la même façon partout sur terre. Certaines races sont plus exposées à l’amidon que d’autres, qui se nourrissent presque uniquement de viande (de mammifère ou de poisson). Les biologistes ont prédit que le nombre de copies du gène AMY2 serait plus élevé chez les races de chien qui sont plus en contact avec une diète contenant de l’amidon semblable à celle des humains. Ils ont comparé des races de chien qui ne consomment peu ou pas d’amidon et qui ont une diète contenant des mammifères terrestres et marins et du poisson (le Malamute d’Alaska, le Shiba Inu japonais et le Husky sibérien) avec des races connues pour avoir une diète composée en plus grande partie d’amidon, car ils sont traditionnellement associés à des populations vivant d’agriculture de céréales (Le Shar Pei chinois et le Pékinois)(figure 21.6). Ils ont utilisé un échantillonnage de cellules de la muqueuse buccale des chiens pour estimer le nombre de copies du gène dans leur génome, une méthode non invasive qui leur a permis d’avoir une grande taille d’échantillon pour chaque race. Ils ont démontré que le nombre de copies du gène AMY2 variait avec l’apport alimentaire en amidon (figure 21.6). Les chiens avec des régimes riches en amidon avaient en moyenne 10 copies du gène comparativement aux chiens se nourrissant principalement de viande qui en avait 7 en moyenne. Comme chez l’humain, les biologistes ont démontré que les individus avec un nombre élevé de copies avaient le même type de diète mais n’étaient pas plus proches génétiquement lorsque la distance génétique était estimée avec des marqueurs neutres. Cette déconnexion entre le cladogramme qui illustre la distance génétique et le nombre de copies à un gène spécifique suggère que la sélection naturelle a agi spécifiquement sur le phénotype qui est associé avec un grand nombre de copies du gène AMY2B. Par exemple, bien que le cladogramme révèle que le Shar Pei est plus étroitement apparenté au Malamute d’Alaska, au Husky de Sibérie et aux chiens japonais, le Shar Pei a un nombre moyen de copies plus élevé, plus semblable à celui des Pékinois [15].
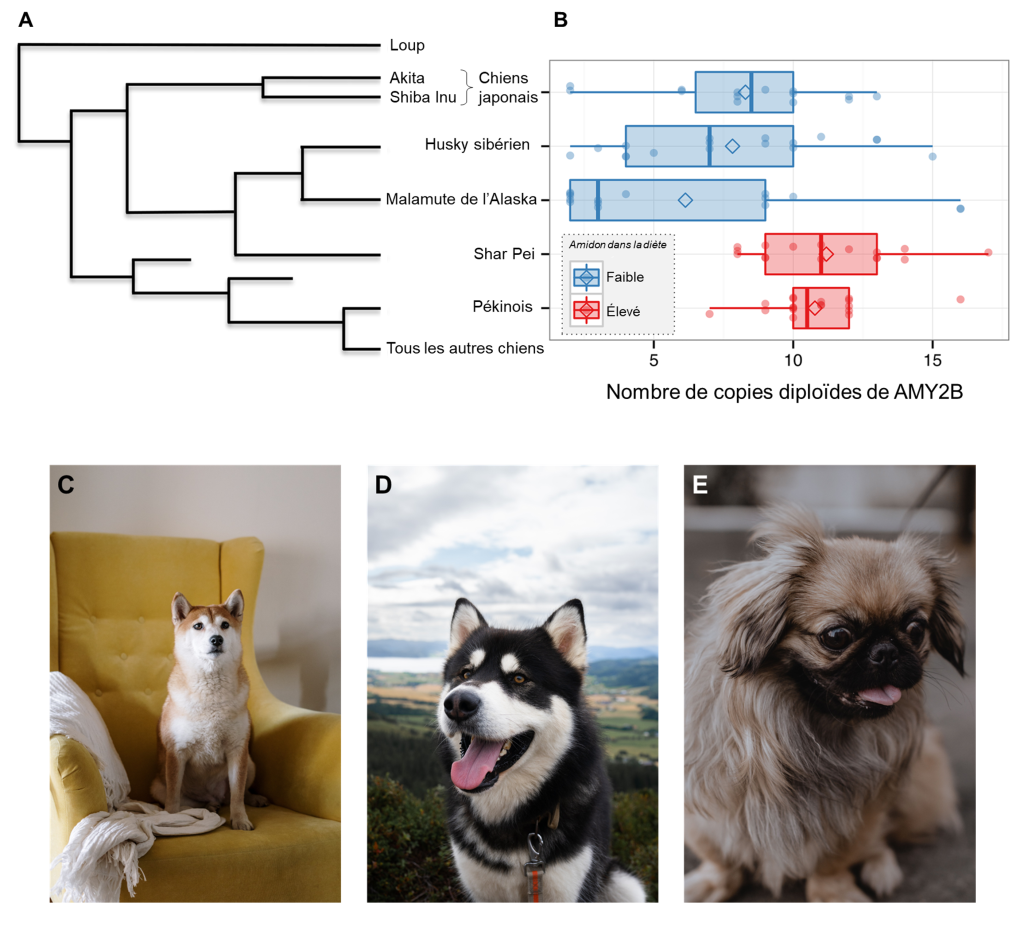
2.7 Évolution indépendante de la duplication des gènes codant pour l’amylase et du nombre de copies chez les mammifères domestiqués
Les chiens ne sont pas les seuls animaux qui vivent à proximité des humains. Il y a bien sûr les autres animaux domestiqués comme les animaux de la ferme, mais aussi les espèces qui utilisent les milieux de vie des humains comme source de nourriture, tels que les souris et les rats. Des études avaient déjà démontré que les souris et les porcs ont, comme les humains, deux versions du gène AMY (AMY2 qui code pour l’amylase sécrétée par le pancréas et AMY1 qui code pour une amylase salivaire). Des biologistes ont démontré que le génome d’une souris de laboratoire a plus de deux copies du gène AMY2 [19] et que pour différentes races de porcs, les différents gènes AMY présents dans leurs génomes ont tous un nombre élevé de copies, avec entre 8 et 21 copies [20].
Des biologistes se sont intéressés à plusieurs autres mammifères vivant à proximité des humains afin de déterminer lesquels ont les deux paralogues AMY1 et AMY2 comme la souris, le porc et l’humain et si ces espèces ont plus de deux copies de chaque gène. Pourquoi se sont-ils intéressés à cette question? Si au fil de l’évolution les différentes espèces qui ont une augmentation de l’amidon dans leur diète ont aussi une augmentation du nombre de copies des gènes qui codent pour l’amylase (soit une duplication qui ajoute l’amylase salivaire, soit une duplication qui augmente le nombre de copies d’un gène en particulier), cette évolution convergente supporterait l’hypothèse que c’est une adaptation liée à la pression de sélection d’obtenir des sources de glucides. Ils ont étudié le génome de 46 espèces de mammifères représentant des espèces avec une diète contenant une haute proportion d’amidon et d’autres une faible proportion et ont inclus des espèces sauvages, domestiques et des espèces associées aux humains (« commensales »). Ces espèces incluaient plusieurs primates (babouin, bonobos, capucin, chimpanzé, gorille, ouistiti, orang-outang, macaque, humain), le mouflon d’Amérique et le bison, le chat et le puma, le loup et le chien, la vache, le cheval, la chèvre, le mouton et le yak, le cochon et le sanglier, l’ours noir et l’ours polaire, le zèbre, la souris et le rat ainsi que divers rongeurs sauvages.
Ils ont observé une augmentation du nombre de copies (duplication de gène menant à des paralogues) et une variation du nombre de copies entre les individus (variants) chez plusieurs espèces (figure 21.7A). Ils ont utilisé une reconstruction phylogénétique pour déterminer si cette duplication menant aux deux paralogues AMY2 et AMY1 était présente chez l’ancêtre des mammifères et aurait été perdue chez certains groupes, ou si elle est apparue indépendamment plusieurs fois au cours de l’évolution (évolution convergente). Cet « arbre de gène » est créé en utilisant les séquences des gènes AMY de chaque espèce et montre lesquelles sont les plus similaires entre elles. Si tous les gènes AMY1 étaient regroupés sur une branche et que toutes les séquences AMY2 des différentes espèces étaient sur une autre branche, cela indiquerait que la duplication a eu lieu dans l’ancêtre commun, avant que ces espèces ne divergent, et que les copies ont été conservées au fil du temps évolutif. Si les gènes AMY1 et AMY2 et les différentes copies d’un gène donné se regroupent ensemble pour une espèce, cela supporte l’hypothèse que les duplications ont eu lieu indépendamment dans chaque espèce. Leur analyse supporte l’hypothèse d’une évolution convergente, car les gènes codant pour l’amylase au sein d’une espèce donnée étaient plus similaires en séquence les uns aux autres qu’ils ne l’étaient avec ceux des autres espèces (figure 21.7B) [21].
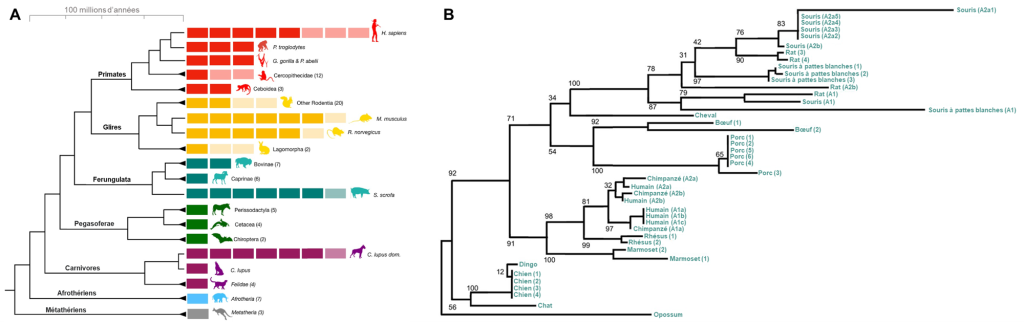
2.8 Association entre la diète, le nombre de copies de gènes codant pour l’amylase et l’amylase salivaire
Les biologistes se sont demandé s’il y avait une association entre le nombre de copies des gènes codant pour l’amylase (AMY1 et AMY2 confondus), l’activité de l’amylase dans les espèces où ils ont pu le mesurer, et la diète décrite dans différents articles scientifiques. Ils ont classé les mammifères en spécialistes (carnivores ou herbivores) et généralistes. Ces derniers ont été subdivisés selon qu’ils consomment de grandes quantités d’amidon (humains, souris, rats bruns et noirs, chiens, porcs et sangliers) ou peu d’amidon. Conformément à leurs prédictions, le nombre de copies des gènes codant pour l’amylase était plus élevé chez les grands consommateurs d’amidon (figure 21.8).
Pour ce qui est de l’activité de l’amylase salivaire, il y avait une différence significative entre les spécialistes et les généralistes, mais pas selon la quantité d’amidon consommé. L’activité était aussi élevée chez les espèces qui consomment un peu d’amidon que celles qui en consomment beaucoup, ce qui suggère selon eux que l’amylase salivaire pourrait avoir une fonction de détecter la présence d’amidon, car cette enzyme le transforme en sucres simples qui peuvent activer les récepteurs du gout sucré directement dans la bouche. Les animaux pourraient donc détecter une source d’énergie élevée, ce qui n’est pas possible si l’amidon n’est pas hydrolysé. Cependant, cela est une hypothèse qui reste à tester. Cette évolution rapide dans de nombreuses espèces suggère que la duplication de gènes et la variation du nombre de copies sont des mécanismes à prendre en compte lors de l’étude de l’évolution d’adaptations [21].
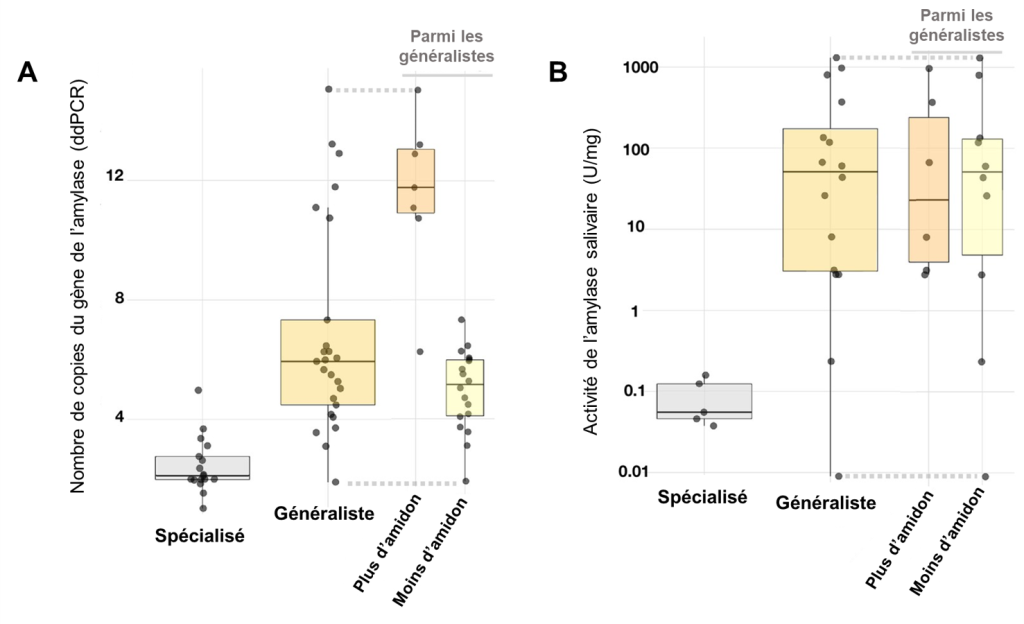
Exercices
Duplication du gène de l’amylase
3. La lactase
L’introduction de la section 3 et les sous-sections 3.1 et 3.2 sont adaptées de [22].
Nous allons maintenant discuter d’une possible adaptation qui découle d’une caractéristique particulière des mammifères : la production de lait pour nourrir nos petits. Le lait contient naturellement non seulement des protéines et des lipides; il contient également des glucides. Plus précisément, le lait contient le sucre lactose. Le lactose est un disaccharide composé d’une molécule de galactose et d’une molécule de glucose (figure 21.9). Le lactose représente entre 2 et 8 % du poids du lait. La quantité exacte de lactose varie à la fois entre les individus et entre les espèces de mammifères.
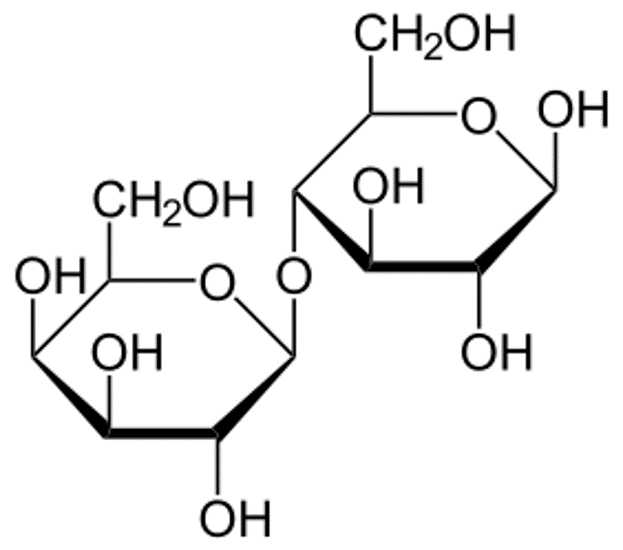
Le lactose du lait doit être décomposé en ses deux sucres composants pour être absorbé par l’intestin grêle. L’enzyme lactase est nécessaire pour ce processus (figure 21.10). Les mammifères naissent presque toujours avec la capacité de synthétiser la lactase. Cela leur permet de digérer facilement le lactose du lait maternel. Cependant, chez la majorité des nourrissons, la synthèse de lactase commence à décliner avec l’âge (vers l’âge de 2 ans chez les humains) et de moins en moins de lactase est produite tout au long de l’enfance.
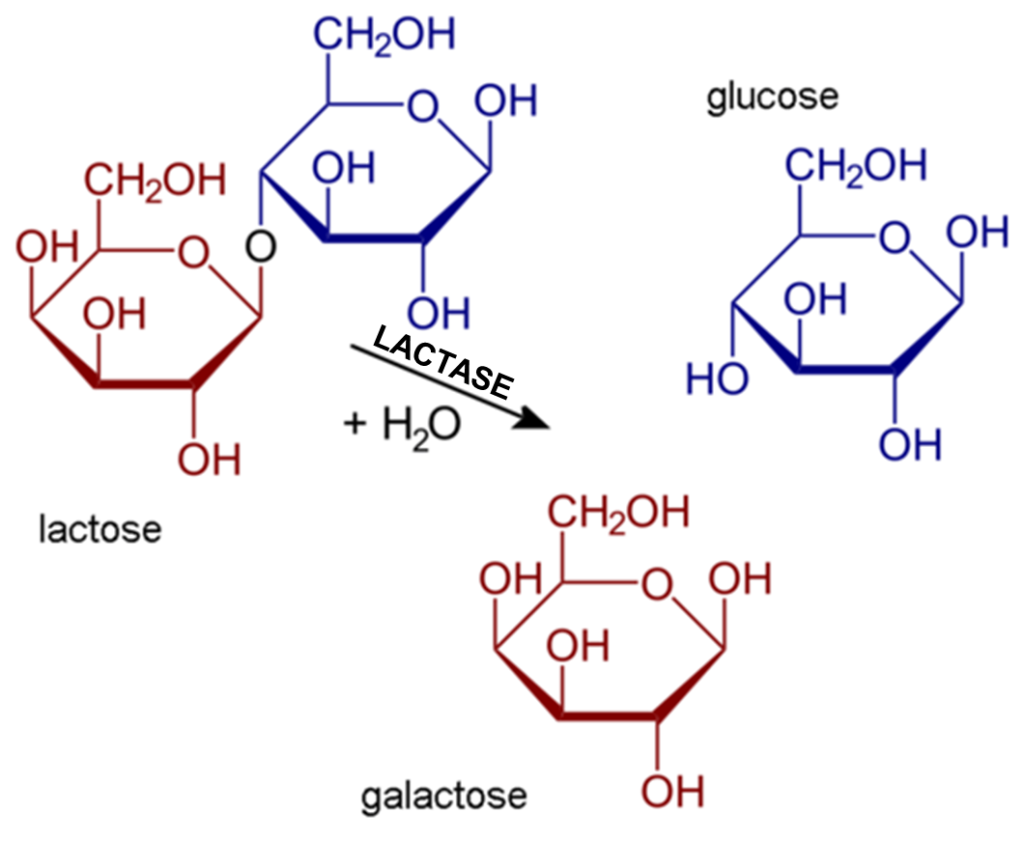
L’intolérance au lactose est l’incapacité des jeunes qui ne sont plus des nourrissons et des mammifères adultes à digérer le lactose dans le lait. Chez l’humain, les personnes intolérantes au lactose peuvent être capables de boire de petites quantités de lait sans aucun problème, mais si elles essaient d’en consommer de plus grandes quantités, elles risquent de subir des effets indésirables. Leurs symptômes résultent de l’incapacité de l’intestin grêle à digérer et à absorber le lactose, de sorte que le lactose est transmis au gros intestin, où les bactéries intestinales normales commencent à le décomposer par le processus de fermentation. Ce processus libère des gaz et provoque les symptômes d’intolérance au lactose. Par exemple, les personnes intolérantes peuvent avoir des ballonnements et des crampes abdominales, des flatulences, de la diarrhée, des nausées et des vomissements. Les symptômes peuvent survenir une demi-heure à deux heures après la consommation de lait et s’aggravent généralement lorsque la quantité de lait consommée est plus importante.
3.1 La persistance de la lactase à l’âge adulte chez les humains
L’intolérance au lactose après le stade de lactation est la condition ancestrale de toutes les espèces de mammifères, incluant les humains. Les humains sont la seule espèce connue de mammifère dans laquelle certains adultes sont capables de digérer le lactose. Cette capacité est due à la persistance de la lactase, qui signifie que l’enzyme continue d’être produite à l’âge adulte.
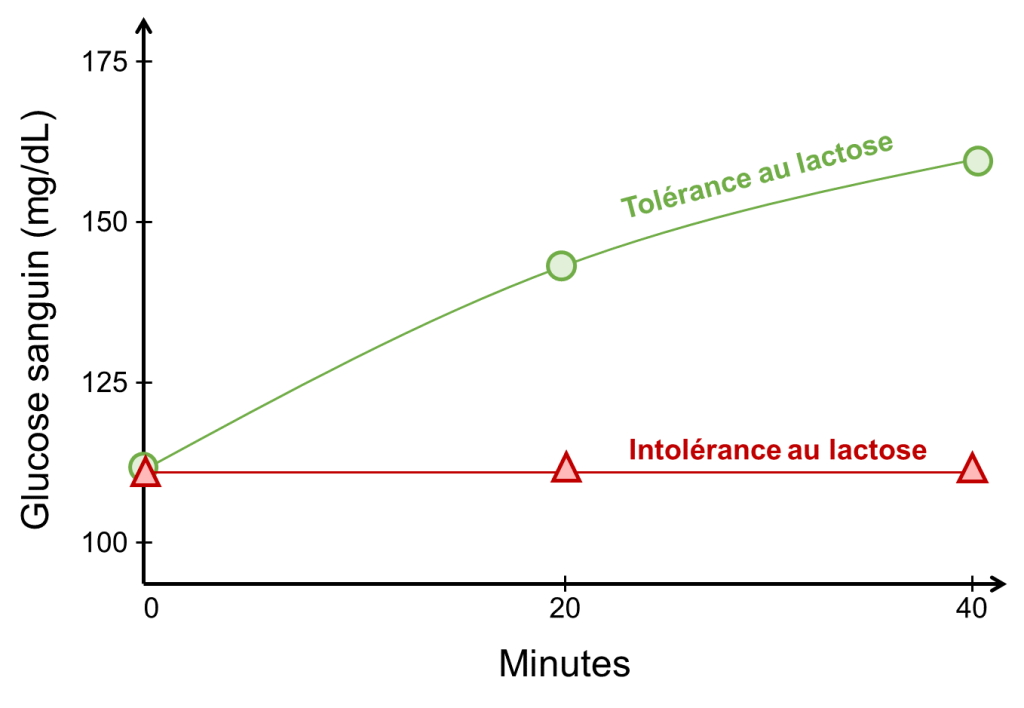
Pour déterminer si un adulte possède une enzyme lactase persistante, on peut mesurer son taux de glucose sanguin avant et après l’ingestion de lait. On prend ensuite des mesures après 20 et 40 minutes par exemple, pour laisser le temps au lactose d’être digéré. Si la lactase est présente, elle va décomposer le lactose en glucose et galactose et le glucose sanguin va donc augmenter. Si la personne n’a pas de persistance de la lactase, on aura une pente nulle et un sujet avec des symptômes bien désagréables! (figure 21.11).
3.2 Les populations humaines ont différents niveaux de persistance de la lactase
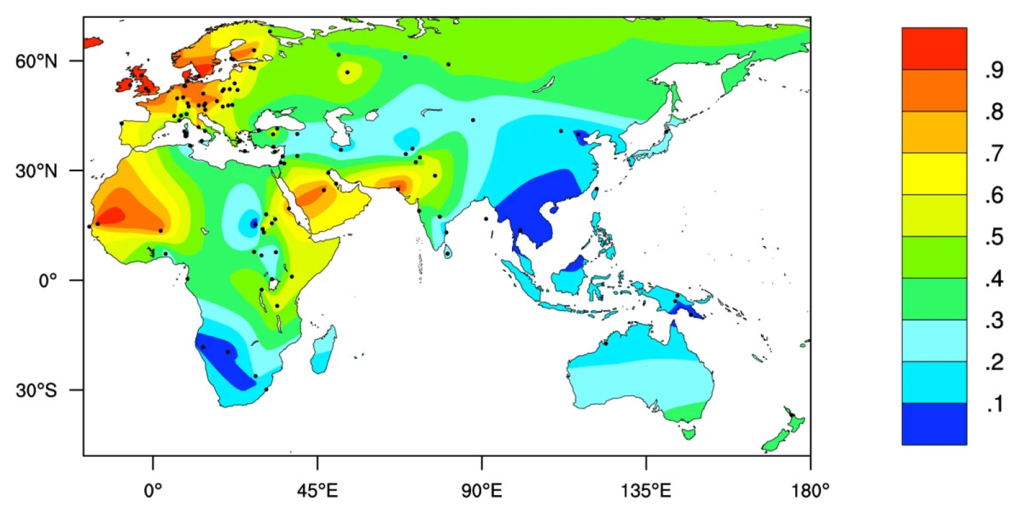
Dans l’ensemble, on estime que 35 % de la population humaine mondiale a une enzyme lactase persistante à l’âge adulte. Cette persistance de l’activité de l’enzyme qui digère le lactose varie cependant énormément entre les populations (figure 21.12). La persistance de la lactase à l’âge adulte se trouve à des fréquences modérées à élevées chez les Européens et certaines populations d’Afrique, du Moyen-Orient et d’Asie du Sud. Par contre, elle est plus rare dans d’autres régions, tel qu’on peut le voir sur la carte (figure 21.12).
3.3 Adaptation au lactose dans la diète à l’âge adulte chez l’humain
Nous pouvons nous demander si ce trait de persistance de la lactase à l’âge adulte est une adaptation. Afin de déterminer si c’est le cas, nous avons besoin de deux informations. Premièrement, nous devons savoir si la variation de phénotype de persistance entre les individus résulte de la variation génétique (c.a.d que ce trait a une héritabilité élevée), ou si cette persistance représente de la plasticité phénotypique. Deuxièmement, nous devons savoir si ce phénotype augmente le fitness comparativement aux autres formes de ce trait, dans l’environnement où on le retrouve.
3.4 Utilisation de pedigree humain pour déterminer le mode d’hérédité
Avant de montrer que la persistance de la lactase est une adaptation, il faut vérifier que les différences de tolérances soient dues à de la variation génétique. Si le trait est bénéfique et améliore la reproduction et la survie (il y a de la sélection naturelle) mais ne peut pas être transmis aux descendants (l’héritabilité est de zéro), nous ne verrons pas de réponse à la sélection dans la prochaine génération et il n’y aura pas d’évolution.
La chose la plus simple à tester est de vérifier si la variation de la tolérance résulte de la plasticité face à l’environnement. Par exemple, si le fait de boire du lait permet de demeurer tolérant au lactose, on serait devant un cas de plasticité. Selon cette hypothèse, les gens exposés continuellement au lactose pourraient demeurer tolérants et c’est par manque d’exposition au lactose que la lactase déclinerait. Cependant, différentes données montrent que ce n’est pas le cas.
Comme premier pas pour vérifier si la variation dans la persistance de la lactase est liée à des différences génétiques, nous pouvons vérifier si les parents et les enfants ont le même niveau de tolérance au lactose. Nous allons présenter les données de phénotype dans un schéma qu’on appelle un pedigree.
Le paragraphe suivant est adapté de [27].
La transmission d’un trait à travers les générations peut être montrée visuellement à l’aide d’un arbre généalogique sur lequel on superpose la présence ou l’absence d’un phénotype, schéma qu’on appelle un pedigree (figure 21.13). Les formes carrées représentent les mâles (les hommes chez les humains); les cercles représentent les femelles (les femmes chez les humains). Les formes remplies sont des individus qui ont le phénotype étudié dans le pedigree. Deux individus reliés par une ligne horizontale entre eux sont les parents des individus qui sont reliés par des lignes verticales en dessous d’eux. Les frères et sœurs sont généralement affichés par ordre de naissance avec l’ainé à gauche.
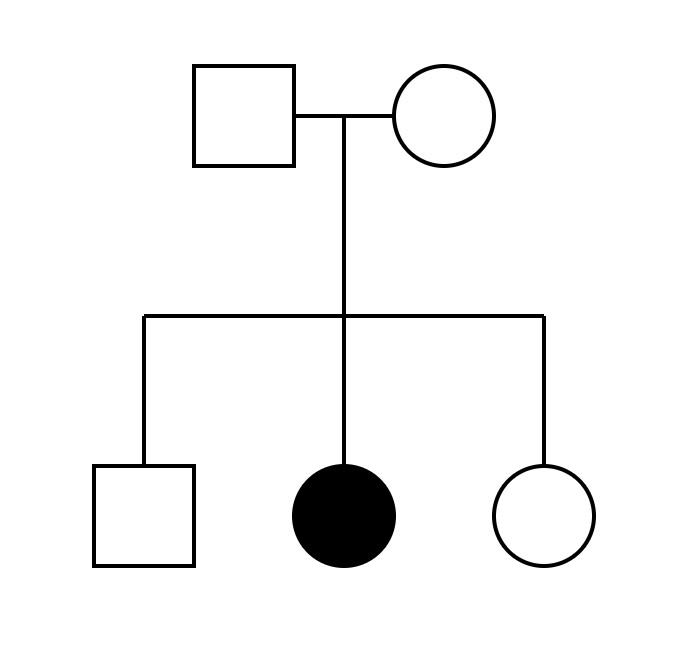
Un pedigree est donc une façon de représenter les relations entre les gens qui sont apparentés et de suivre la présence d’un phénotype qui nous intéresse. On peut souvent voir à gauche du pedigree des chiffres romains qui indiquent la génération et des chiffres arabes sous les individus pour pouvoir y référer dans un texte. Nous voulons suivre qui est tolérant au lactose à l’âge adulte chez des familles, pour voir si le phénotype des parents prédit le phénotype de leurs descendants.
Les pedigrees
Amoeba Sisters. (2017). Pedigrees [Vidéo]. Youtube. https://www.youtube.com/watch?v=Gd09V2AkZv4
3.4.1 Déterminer si un trait est dominant ou récessif à l’aide d’un pedigree
Des biologistes ont étudié des données sur des Finlandais sur 5 générations afin de créer un pedigree de l’intolérance au lactose. En effet, comme dans cette population la majorité des gens sont tolérants au lactose, le phénotype plus rare à étudier est le fait d’être intolérant. Ainsi, selon le code d’un pedigree, on cherche à suivre le phénotype d’intolérance au lactose et on va le colorer en noir. Ils ont observé que des parents intolérants ont toujours des enfants intolérants et que des parents tolérants ont parfois des enfants intolérants. Finalement, ils ont observé des individus tolérants ou intolérants dans les deux sexes.
On se souvient qu’un allèle dominant résulte en un phénotype aussitôt que quelqu’un a une copie de l’allèle et qu’un allèle récessif ne résulte en un phénotype que si la personne a les deux copies de cet allèle. Donc si l’intolérance au lactose est un trait dominant, un hétérozygote serait intolérant alors que si l’intolérance au lactose est un trait récessif, il faut être homozygote pour les deux allèles récessifs pour avoir le phénotype intolérant. Le patron de distribution du phénotype entre les parents et les enfants suggère qu’il faut avoir les deux copies de l’allèle « intolérant » pour exprimer le phénotype d’intolérance au lactose (figure 21.14). En se basant sur les données de ce pedigree, les biologistes ont déterminé que le trait de l’intolérance au lactose à l’âge adulte est donc récessif. On sait que le trait de l’intolérance au lactose n’est pas lié à un chromosome sexuel, car si c’était le cas, on aurait un sexe qui l’exprimerait beaucoup plus souvent [29].

Exercices
Analyse de pedigree
Les sous-sections 3.4.2 et 3.4.3 sont adaptées de [31].
3.4.2 Mutation dans la séquence codante du gène codant pour la lactase
Des chercheurs ont décidé de vérifier s’il existe une mutation dans la séquence du gène codant pour la lactase qui serait associée à la persistance de la lactase. Chez les humains, le gène qui code pour la lactase est appelé LCT et se trouve sur le chromosome 2. Parce que le chromosome 2 est un autosome, tout le monde a deux copies du gène LCT, une sur son chromosome 2 maternel et une sur son chromosome 2 paternel. Le gène LCT a une longueur d’environ 55 kilobases. Il est exprimé spécifiquement dans les cellules épithéliales de l’intestin et pas dans d’autres tissus. Chez les personnes tolérantes au lactose, il est exprimé du début de la vie jusqu’à l’âge adulte. Chez les personnes intolérantes (comme chez la plupart des mammifères), il cesse d’être exprimé chez les enfants au-delà du stade de lactation. La lactase est une protéine de la membrane plasmique. Comme beaucoup d’autres protéines, elle est synthétisée par des ribosomes dans le réticulum endoplasmique rugueux, modifiée par des enzymes dans l’appareil de Golgi, et finalement déplacée dans une vésicule de transport jusqu’à la surface de la cellule (figure 21.15). La protéine mature est longue de 1060 acides aminés. La majeure partie de la protéine, y compris son site actif, se trouve à l’extérieur de la cellule. Elle possède un seul domaine transmembranaire qui l’ancre dans la membrane. Il existe également une petite partie cytosolique de la protéine à son extrémité carboxyle.
Comment pouvons-nous savoir si la persistance de la lactase est associée à une mutation spécifique dans le gène LCT? On peut par exemple obtenir de l’ADN d’humains adultes qui sont tolérants au lactose et d’autres qui sont intolérants et séquencer la région de l’ADN qui contient le gène qui code pour l’enzyme lactase. Si on trouve des mutations dans la séquence de nucléotides qui se retrouvent uniquement dans un groupe, on peut vérifier si cela affecte la séquence en acide aminé (substitutions non synonymes) et étudier comment cela affecte la forme et la fonction de la protéine. Dans le cas qui nous intéresse, pour faire une histoire courte, les biologistes n’ont pas trouvé de différences dans la séquence codante du gène de la lactase. La protéine qui forme la lactase a la même forme chez tous les humains, qu’ils soient tolérants au lactose à l’âge adulte ou non.
3.4.3 Mutation dans la séquence régulatrice du gène codant pour la lactase
Les biologistes ont émis l’hypothèse que la variation génétique affectant la persistance de la lactase devrait être dans la séquence régulatrice du gène, c’est-à-dire la séquence d’ADN qui contrôle l’expression du gène qui code pour l’enzyme. Si le gène est plus exprimé, il y a plus de lactase. Si le gène n’est pas exprimé, il n’y a pas de lactase. On se rappelle que la distribution de la persistance de la lactase dans la population humaine est plus élevée dans trois points chauds : l’Europe du Nord, l’Afrique de l’Est, le Moyen-Orient et l’Asie du Sud (figure 21.12 plus haut). Après de nombreuses recherches en comparant le génome d’individus tolérants et intolérants, les scientifiques ont découvert que dans chaque cas, il y avait une seule mutation responsable. Dans la population européenne, une substitution de paires de bases CG à TA a créé l’allèle qui confère la persistance de la lactase. La raison pour laquelle il a fallu si longtemps pour la trouver est que la mutation était loin de la région transcrite, 13 910 paires de bases en amont! Les allèles de persistance de la lactase dans les autres populations sont formés de différentes substitutions de nucléotides très proches de celle trouvée en Europe (Figure 21.16). Dans les populations africaines, il s’agit d’une substitution TA -> GC à -13 915 pb, tandis qu’au Moyen-Orient et en Asie du Sud, il s’agit d’une substitution CG à GC à 13 907 pb en amont du gène LCT.
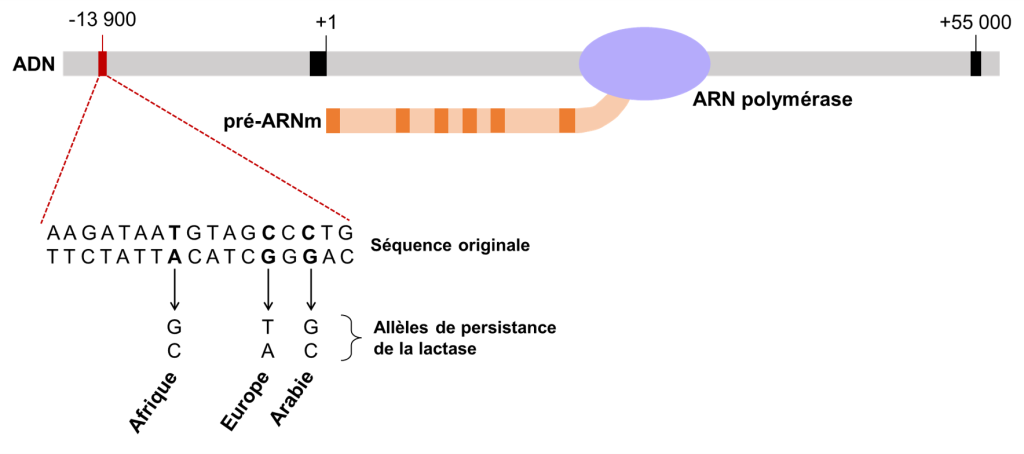
3.4.4 Quantification de l’effet fonctionnel de la mutation
Ce n’est pas parce qu’on trouve une mutation associée à un trait (ici la persistance de la lactase et le nucléotide T à la position -13 910 chez les européens) qu’on peut automatiquement dire que c’est cette mutation qui cause le phénotype. Il est nécessaire de faire une analyse fonctionnelle où on teste l’effet de cette mutation sur le fonctionnement de la cellule. La région d’ADN en amont du gène LCT où se trouve la mutation est une séquence régulatrice qu’on appelle une zone amplificatrice (un enhancer en anglais). Cette portion d’ADN est un site de liaison pour un facteur de transcription positif (ou « activateur »), c’est-à-dire une protéine qui se lie aux gènes et les active. Dans le cas présent, le facteur de transcription positif qui se lie à zone amplificatrice et active le gène LCT est une protéine appelée Oct-1.
Des biologistes ont démontré directement que Oct-1 est un activateur pour le gène qui code pour la lactase (LCT) de sorte qu’il augmenterait la transcription du gène LCT. Pour y arriver, ils ont utilisé des lignées cellulaires qui représentent bien le fonctionnement de l’épithélium digestif humain et qui sécrètent différentes enzymes de digestion, dont la lactase, même en culture en laboratoire. Ils ont introduit la mutation T des individus tolérants au lactose en Europe au site -13 910. Ils ont mesuré le niveau d’expression du gène de la lactase chez une lignée cellulaire non modifiée et la lignée avec cette mutation. Ils ont trouvé des effets positifs significatifs sur les niveaux d’expression, confirmant le lien de cause à effet [33].
Fonctionnement d’une zone amplificatrice et d’un facteur de transcription positif
Chantal Proulx. (2012). Régulation génique [Vidéo]. Youtube. https://youtu.be/eEe-nkyjmqw?si=MOigytcA07GOqYCz&t=26
3.5 Évolution convergente de la persistance de la lactase
Nous avons vu qu’il existe plusieurs allèles différents qui confèrent la persistance de la lactase à l’âge adulte dans différentes populations humaines. Ce phénotype semble être apparu plusieurs fois de manière indépendante dans les populations humaines. Les mutations elles-mêmes sont apparues par hasard, comme toutes les mutations, mais leur montée en fréquence en parallèle dans différentes populations suggère qu’une pression de sélection élevée a été exercée sur le phénotype de persistance de la lactase que cette mutation confère (ou contre le phénotype d’intolérance au lactose lié à la séquence d’ADN originale) et que ce trait pourrait être une adaptation chez les humains. Il faudrait maintenant déterminer l’agent de sélection, ou les agents de sélection, car on ne peut pas présumer que l’augmentation en fréquence des personnes tolérantes au lactose à l’âge adulte a la même cause dans toutes les populations humaines et a une seule cause dans une population donnée.
3.5.1 Lien entre l’évolution de la persistance de la lactase et les développements socioculturels chez l’humain.
La recherche sur l’évolution de la persistance de la lactase chez l’humain est interdisciplinaire. En effet, elle demande de connecter des informations venant de toutes sortes de niveaux d’organisation et de connecter les sciences de la vie avec les sciences humaines. Par exemple, un article scientifique de 2023 sur ce sujet compte 108 auteurs provenant de 20 pays et inclut des disciplines aussi variées que la chimie, l’épidémiologie, les sciences médicales, la génétique et la paléogénétique, l’évolution, l’archéologie et l’anthropologie, l’étude de la préhistoire et l’ethnologie, les sciences animales, la muséologie, l’histoire, la géographie, la géologie et la physique nucléaire. Ces scientifiques tentent de connecter l’évolution du génome des populations humaines et leurs effets sur leur phénotype avec l’habitat des humains vivant il y a entre 9000 et 2000 ans en Europe. De nombreuses avancées culturelles chez l’humain datent de cette période, incluant l’utilisation du lait provenant d’autres espèces comme source de nourriture et l’élevage puis la domestication d’espèces de mammifères produisant du lait [34].
L’hypothèse la plus simple, et celle qui a été enseignée durant plusieurs décennies, est que l’arrivée de la consommation de lait chez les humains adultes a été l’agent de sélection menant à l’augmentation de la fréquence du génotype résultant en persistance de la lactase. Cette hypothèse suppose que les personnes qui avaient l’allèle qui confère la persistance de la lactase (que nous nommerons l’allèle « PL ») pouvaient obtenir plus d’énergie du lait que les personnes qui ne l’avaient pas, ce qui entrainait des différences de croissance, de fréquence de reproduction et de survie, qui menaient à une augmentation de rejetons portant l’allèle PL (qui rappelez-vous, est dominant). Si tel est le cas, on peut prédire que les traces archéologiques de consommation de lait seraient étroitement associées dans le temps avec l’augmentation de la fréquence de personnes tolérantes au lactose à l’âge adulte, que l’on peut estimer en quantifiant la fréquence de l’allèle PL dans une population.
L’étude menée par les 108 scientifiques présente des données permettant de tester cette prédiction. Ces données ont été rendues possibles grâce à la combinaison de paléogénomique utilisant l’ADN ancien d’humain et des méthodes de datation et de caractérisation des résidus de lipides dans les contenants de poteries trouvées par des archéologues dans différents sites en Europe, allant d’il y a 7000 ans avant l’ère commune jusqu’à l’an 1000 de notre ère. Cette étude ne supporte pas la prédiction que l’arrivée de la consommation de lait a coïncidé avec l’augmentation de la tolérance au lactose, au moins pour l’Europe et l’allèle PL au site « -13 910 ». En effet, l’analyse des sites de fouilles archéologiques montre que la consommation de lait par les humains a commencé en Eurasie en 7000 AEC (avant l’ère commune) et que l’art de la laiterie était déjà répandu en 5000 AEC. Cependant, les études de génomique et de l’ADN ancien ont permis de déterminer que le patron d’augmentation de la fréquence de l’allèle PL au fil des générations dans les populations humaines de l’Eurasie date plutôt de 3000 ans AEC. Il y a donc une période de 2000 ans où la très grande majorité des humains vivant en Europe n’avaient pas l’allèle conférant la persistance de la lactase mais consommaient probablement du lait à l’âge adulte.
Ce groupe de scientifiques suggère que l’avantage de pouvoir digérer le lait à l’âge adulte, et le désavantage de souffrir d’effets secondaires digestifs négatifs pour les personnes qui n’étaient pas tolérantes au lactose, ont augmenté durant des périodes spécifiques de l’histoire de l’humanité dans cette région. En effet, il y a bel et bien un signal de balayage sélectif dans l’augmentation de la fréquence de l’allèle PL dans des populations comme le territoire du Royaume-Uni actuel, où jusqu’à 75 % des gens ont l’allèle PL de nos jours. Ils proposent deux scénarios qui peuvent avoir eu lieux seuls ou en combinaison, où les gens qui ne digéraient pas le lactose étaient désavantagés et avaient un fitness moindre, ce qui a mené à une diminution de l’allèle original associé à l’intolérance au lactose : 1) lors d’épisodes de pénuries de nourriture et 2) lorsqu’ils étaient plus exposés à des pathogènes animaux causant des zoonoses. Il faut noter que le scénario de l’augmentation de l’exposition à des pathogènes pourrait se matérialiser lorsqu’il y a une famine et que plus d’animaux sauvages entrent dans la diète pour pallier les manques de nourriture, ou au contraire lorsque les humains vivent dans des communautés plus denses et à proximité d’animaux domestiqués, ou ces animaux et les autres humains leur transmette des maladies. Pour tous ces scénarios indépendants ou en combinaison, la survie d’une personne devient extrêmement liée à l’apport énergétique des produits laitiers et cet apport est alors significativement différent entre les personnes ayant l’allèle PL et celles souffrant de troubles digestifs liés à l’incapacité de digérer le lactose. Ainsi, dans le scénario de famine, les produits laitiers sont plus consommés pour pallier le manque de nourriture, mais la diarrhée induite par le lactose chez les gens qui ne produisent pas de lactase peut devenir mortelle chez les personnes souffrant de malnutrition sévère. Cela résulte en une pression de sélection élevée pour les gens pouvant digérer le lactose, qui ont nécessairement au moins un allèle PL. Dans le scénario d’exposition aux pathogènes, l’incapacité de digérer le lactose créé des problèmes digestifs chroniques et des changements au microbiome intestinal qui rendent propices des effets plus sévères des maladies liées aux pathogènes. Encore une fois, les personnes ne souffrant pas de problèmes chroniques (ayant un allèle PL) survivent mieux et laissent plus de descendance [34, 35].
Cette étude à grande échelle laisse entrevoir qu’il y a plus qu’un agent de sélection qui a mené à l’évolution de ce trait monogénique unique aux humains qu’est la persistance de la lactase à l’âge adulte pour les populations européennes. Le même type d’étude doit maintenant être fait pour comprendre ce qui s’est passé dans les populations africaines qui ont des allèles différents qui confèrent le même phénotype de tolérance au lactose.
Cas appliqué
Texte adapté de [36].
Des biologistes ont analysé une expérience d’évolution expérimentale baptisée « l’expérience Longshanks » (qu’on peut traduire par « longs jarrets »), pour explorer comment le génome d’un animal change lorsqu’un trait est sous une forte pression de sélection. Pendant cinq ans, deux lignées indépendantes de souris ont été sélectionnées pour avoir des tibias plus longs (figure 21.17, panneau A). À chaque génération, les souris ont été mesurées et celles qui avaient le tibia le plus long par rapport à leur masse corporelle ont été sélectionnées pour se reproduire. Les biologistes évolutifs ont analysé les données génétiques des souris de la génération 0 qui ont servi à démarrer l’expérience et celles sélectionnées pendant 17 générations, afin de découvrir quels types de changement dans le génome peuvent être reliés avec l’augmentation de 13 % de la longueur des pattes observée chez les souris sélectionnées (figure 21.17 panneau B et C).
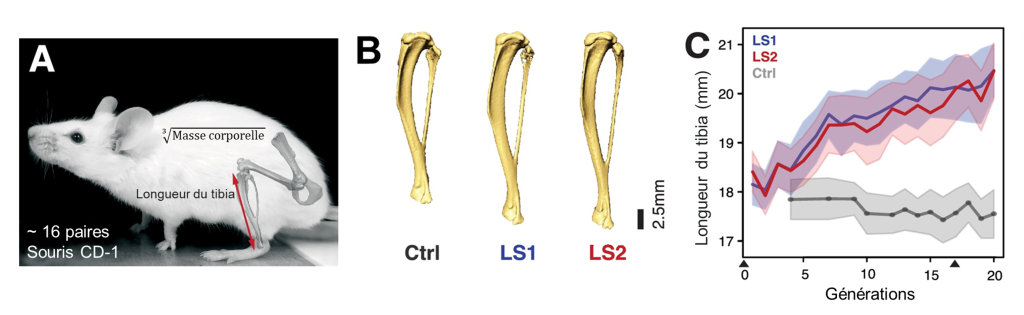
Cette analyse de génomique a révélé de nombreux gènes agissant de concert pour augmenter la longueur du tibia. C’est un donc un trait polygénique. Le gène ayant de loin l’effet le plus important dans les deux lignées sélectionnées était un gène clé du développement appelé Nkx3-2. Les mutations de ce gène provoquent chez l’humain une maladie appelée dysplasie spondylo-mégaépiphysaire-métaphysaire, qui donne des membres longs et un tronc court. Bien que l’inactivation complète de ce gène chez la souris soit mortelle, parmi le groupe fondateur de souris participant à l’expérience Longshanks se trouvait des mutations dans la région génomique proche du gène Nkx3-2. Les individus avec ces mutations expriment le gène Nkx3-2 dans tous les tissus attendus, à l’exception des pattes. Cette région du génome contient une séquence régulatrice. En effet, l’analyse du génome a montré que les polymorphismes à des nucléotides simples chez les souris aux longs tibias étaient dans la séquence de deux activateurs spécifiques qui contrôlent l’expression du gène Nkx3-2 dans les pattes et le tronc. Ces mutations désactivent les activateurs, ce qui fait que le gène Nkx3-2 n’est pas exprimé dans les pattes. Comme la protéine codée par Nkx3-2 agit pour réprimer le développement des os longs et du cartilage, si on désactive un répresseur, on obtient un plus long tibia! Les biologistes ont vérifié expérimentalement que c’est effectivement la mutation spécifique dans l’activateur qui résulte en la perte d’expression de Nkx3-2. Ils n’ont trouvé aucun changement d’acide aminé dans la séquence codante du gène qui pourrait avoir un impact sur la fonction de la protéine.
Résumé
Le système digestif comporte plusieurs enzymes essentielles à son fonctionnement. Deux enzymes sont des exemples très probables d’adaptations chez les humains liées à la niche alimentaire. Le fait d’avoir plusieurs copies du gène codant pour l’amylase résulte en une plus grande quantité de l’enzyme, ce qui permettait de digérer plus d’amidon dans la diète. Cette variation du nombre de copies se retrouve chez les humains et a aussi évolué de façon indépendante chez des animaux domestiqués et des espèces commensales comme les souris. Les humains sont les seuls mammifères connus qui ont une enzyme lactase active à l’âge adulte. Ce phénotype se retrouve chez différentes populations humaines. On peut étudier la transmission de la tolérance au lactose à l’aide d’un pedigree, ce qui a permis de démontrer que c’est un trait dominant autosomal. Une mutation dans la séquence régulatrice fait que l’enzyme lactase continue à être exprimée chez certains humains adultes dans une phase du développement où elle n’est pas active chez les autres espèces de mammifères. Il y a un avantage à digérer le lactose à l’âge adulte quand on a accès à une source de lait qui n’est pas notre mère. La persistance de la lactase a évolué de façon indépendante dans trois populations humaines et est due à des mutations différentes dans la même région du génome.
Activité de révision
Exercices
Révision sur l’évolution de la diète
Bibliographie
- Biga, L.M., S. Bronson, S. Dawson, A. Harwell, R. Hopkins, J. Kaufmann, M. LeMaster, P. Matern, K. Morrison-Graham, K. Oja, D. Quick, J. Runyeon, O. OERU, and OpenStax (2019). Chemical Digestion and Absorption: A Closer Look, in Anatomy & Physiology [CC-BY-SA]. Oregon State University. https://open.oregonstate.education/aandp/chapter/23-7-chemical-digestion-and-absorption-a-closer-look/.
- Betts, J.G., P. Desaix, E. Johnson, J.E. Johnson, O. Korol, D. Kruse, B. Poe, J.A. Wise, M. Womble, and K.A. Young (2022). Digestion and Absorption, in Anatomy and Physiology 2e [CC-BY]. https://openstax.org/books/anatomy-and-physiology-2e/pages/23-7-chemical-digestion-and-absorption-a-closer-look.
- Betts, J.G., P. Desaix, E. Johnson, J.E. Johnson, O. Korol, D. Kruse, B. Poe, J.A. Wise, M. Womble, and K.A. Young (2022). Carbohydrate Digestion Flow Chart, in Anatomy and Physiology 2e [CC-BY]. https://openstax.org/books/anatomy-and-physiology-2e/pages/23-7-chemical-digestion-and-absorption-a-closer-look#fig-ch24_07_02.
- Biga, L.M., S. Bronson, S. Dawson, A. Harwell, R. Hopkins, J. Kaufmann, M. LeMaster, P. Matern, K. Morrison-Graham, K. Oja, D. Quick, J. Runyeon, O. OERU, and OpenStax (2019). The Mouth, Pharynx, and Esophagus, in Anatomy & Physiology [CC-BY-SA]. Oregon State University. https://open.oregonstate.education/aandp/chapter/23-3-the-mouth-pharynx-and-esophagus/.
- Crawley, S.W., M.S. Mooseker, and M.J. Tyska (2014). Shaping the intestinal brush border [CC-BY-NC-SA]. J Cell Biol. 207(4): p. 441-51. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4242837/.
- Freeman, J.L., G.H. Perry, L. Feuk, R. Redon, S.A. McCarroll, D.M. Altshuler, H. Aburatani, K.W. Jones, C. Tyler-Smith, M.E. Hurles, N.P. Carter, S.W. Scherer, and C. Lee (2006). Copy number variation: new insights in genome diversity Genome Res. 16(8): p. 949-61.
- Sebat, J., B. Lakshmi, J. Troge, J. Alexander, J. Young, P. Lundin, S. Månér, H. Massa, M. Walker, M. Chi, N. Navin, R. Lucito, J. Healy, J. Hicks, K. Ye, A. Reiner, T.C. Gilliam, B. Trask, N. Patterson, A. Zetterberg, and M. Wigler (2004). Large-scale copy number polymorphism in the human genome Science. 305(5683): p. 525-8.
- Lindsley, D.L., L. Sandler, B.S. Baker, A.T. Carpenter, R.E. Denell, J.C. Hall, P.A. Jacobs, G.L. Miklos, B.K. Davis, R.C. Gethmann, R.W. Hardy, A.H. Steven, M. Miller, H. Nozawa, D.M. Parry, M. Gould-Somero, and M. Gould-Somero (1972). Segmental aneuploidy and the genetic gross structure of the Drosophila genome Genetics. 71(1): p. 157-84.
- BQmUB2012134 (2012). Amylase reaction [CC0]. https://commons.wikimedia.org/wiki/File:Amylase_reaction.png.
- Groot, P.C., M.J. Bleeker, J.C. Pronk, F. Arwert, W.H. Mager, R.J. Planta, A.W. Eriksson, and R.R. Frants (1989). The human alpha-amylase multigene family consists of haplotypes with variable numbers of genes Genomics. 5(1): p. 29-42.
- Perry, G.H., N.J. Dominy, K.G. Claw, A.S. Lee, H. Fiegler, R. Redon, J. Werner, F.A. Villanea, J.L. Mountain, R. Misra, N.P. Carter, C. Lee, and A.C. Stone (2007). Diet and the evolution of human amylase gene copy number variation Nat Genet. 39(10): p. 1256-60.
- Mandel, A.L., C. Peyrot des Gachons, K.L. Plank, S. Alarcon, and P.A.S. Breslin (2010). Individual Differences in AMY1 Gene Copy Number, Salivary α-Amylase Levels, and the Perception of Oral Starch [CC-BY]. PLOS ONE. 5(10): p. e13352. https://doi.org/10.1371/journal.pone.0013352.
- Inchley, C.E., C.D. Larbey, N.A. Shwan, L. Pagani, L. Saag, T. Antão, G. Jacobs, G. Hudjashov, E. Metspalu, M. Mitt, C.A. Eichstaedt, B. Malyarchuk, M. Derenko, J. Wee, S. Abdullah, F.X. Ricaut, M. Mormina, R. Mägi, R. Villems, M. Metspalu, M.K. Jones, J.A. Armour, and T. Kivisild (2016). Selective sweep on human amylase genes postdates the split with Neanderthals Sci Rep. 6: p. 37198.
- Axelsson, E., A. Ratnakumar, M.-L. Arendt, K. Maqbool, M.T. Webster, M. Perloski, O. Liberg, J.M. Arnemo, Å. Hedhammar, and K. Lindblad-Toh (2013). The genomic signature of dog domestication reveals adaptation to a starch-rich diet Nature. 495(7441): p. 360-364. https://doi.org/10.1038/nature11837.
- Reiter, T., E. Jagoda, and T.D. Capellini (2016). Dietary Variation and Evolution of Gene Copy Number among Dog Breeds [CC-BY]. PLOS ONE. 11(2): p. e0148899. https://doi.org/10.1371/journal.pone.0148899.
- cottonbro studio (2020). Shiba Inu on Yellow Armchair [Licence Pexels]. https://www.pexels.com/photo/shiba-inu-on-yellow-armchair-4056462/.
- Daling, T. (2022). A Close-Up Shot of an Alaskan Malamute [Licence Pexels]. https://www.pexels.com/photo/a-close-up-shot-of-an-alaskan-malamute-13574100/.
- Kondrashin, D. (2022). Close-up Shot of Cute Pekingese Dog [Licence Pexels]. https://www.pexels.com/photo/close-up-shot-of-cute-pekingese-dog-12630573/.
- Schibler, U., A.-C. Pittet, R.A. Young, O. Hagenbüchle, M. Tosi, S. Gellman, and P.K. Wellauer (1982). The mouse α-amylase multigene family sequence organization of members expressed in the pancreas, salivary gland and liver Journal of Molecular Biology. 155(3): p. 247-266. https://www.sciencedirect.com/science/article/pii/0022283682900043.
- Paudel, Y., O. Madsen, H.-J. Megens, L.A.F. Frantz, M. Bosse, J.W.M. Bastiaansen, R.P.M.A. Crooijmans, and M.A.M. Groenen (2013). Evolutionary dynamics of copy number variation in pig genomes in the context of adaptation and domestication BMC Genomics. 14(1): p. 449. https://doi.org/10.1186/1471-2164-14-449.
- Pajic, P., P. Pavlidis, K. Dean, L. Neznanova, R.-A. Romano, D. Garneau, E. Daugherity, A. Globig, S. Ruhl, and O. Gokcumen (2019). Independent amylase gene copy number bursts correlate with dietary preferences in mammals [CC-BY-SA]. eLife. 8: p. e44628. https://doi.org/10.7554/eLife.44628.
- Wakim, S. and M. Grewal Adaptation in Humans, in Introductory Biology: Evolutionary and Ecological Perspectives [CC-BY-SA]. https://pressbooks.umn.edu/introbio/chapter/humansadaptation/.
- NEUROtiker (2007). Lactose Haworth [CC0]. https://commons.wikimedia.org/wiki/File:Lactose_Haworth.svg.
- Yikrazuul (2011). Hydrolysis of lactose [CC0]. https://commons.wikimedia.org/wiki/File:Hydrolysis_of_lactose.svg.
- BioInteractive (2014). The Evolution of Lactose Tolerance — HHMI BioInteractive Video https://www.youtube.com/watch?v=MA9boI1qTuk&t=239s.
- Itan, Y., B.L. Jones, C.J.E. Ingram, D.M. Swallow, and M.G. Thomas (2010). A worldwide correlation of lactase persistence phenotype and genotypes [CC-BY]. BMC Evolutionary Biology. 10(1): p. 36. https://doi.org/10.1186/1471-2148-10-36.
- Anderson, C. and L. Bartee (2016). Pedigrees and Punnett Squares, in Mt Hood Community College Biology 102. https://openoregon.pressbooks.pub/mhccbiology102/chapter/simple-inheritance/.
- Anderson, C. and L. Bartee (2016). A simple pedigree, in Mt Hood Community College Biology 102 [CC-BY]. https://openoregon.pressbooks.pub/mhccbiology102/chapter/simple-inheritance/.
- Enattah, N.S., T. Sahi, E. Savilahti, J.D. Terwilliger, L. Peltonen, and I. Järvelä (2002). Identification of a variant associated with adult-type hypolactasia Nature Genetics. 30(2): p. 233-237. https://doi.org/10.1038/ng826.
- Madibc68 (2019). Example of a Classic Autosomal Recessive Pedigree [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Wiki_Drawing_-_Autosomal_Recessive_(2).svg.
- Locke, J. (2017). Eukaryotic Genes : The Human Lactase (LCT) Gene, in « Open Genetics Lectures » textbook for an Introduction to Molecular Genetics and Heredity (BIOL207) [CC0]. Borealis. https://doi.org/10.7939/DVN/XMUPO6.
- Wanes, D., D.M. Husein, and H.Y. Naim (2019). Congenital Lactase Deficiency: Mutations, Functional and Biochemical Implications, and Future Perspectives [CC-BY]. Nutrients. 11(2): p. 461. https://www.mdpi.com/2072-6643/11/2/461.
- Olds, L.C. and E. Sibley (2003). Lactase persistence DNA variant enhances lactase promoter activity in vitro: functional role as a cis regulatory element Human molecular genetics. 12(18): p. 2333-2340.
- Evershed, R.P., G. Davey Smith, M. Roffet-Salque, A. Timpson, Y. Diekmann, M.S. Lyon, L.J.E. Cramp, E. Casanova, J. Smyth, H.L. Whelton, J. Dunne, V. Brychova, L. Šoberl, P. Gerbault, R.E. Gillis, V. Heyd, E. Johnson, I. Kendall, K. Manning, A. Marciniak, A.K. Outram, J.D. Vigne, S. Shennan, A. Bevan, S. Colledge, L. Allason-Jones, L. Amkreutz, A. Anders, R.M. Arbogast, A. Bălăşescu, E. Bánffy, A. Barclay, A. Behrens, P. Bogucki, Á. Carrancho Alonso, J.M. Carretero, N. Cavanagh, E. Claßen, H. Collado Giraldo, M. Conrad, P. Csengeri, L. Czerniak, M. Dębiec, A. Denaire, L. Domboróczki, C. Donald, J. Ebert, C. Evans, M. Francés-Negro, D. Gronenborn, F. Haack, M. Halle, C. Hamon, R. Hülshoff, M. Ilett, E. Iriarte, J. Jakucs, C. Jeunesse, M. Johnson, A.M. Jones, N. Karul, D. Kiosak, N. Kotova, R. Krause, S. Kretschmer, M. Krüger, P. Lefranc, O. Lelong, E. Lenneis, A. Logvin, F. Lüth, T. Marton, J. Marley, R. Mortimer, L. Oosterbeek, K. Oross, J. Pavúk, J. Pechtl, P. Pétrequin, J. Pollard, R. Pollard, D. Powlesland, J. Pyzel, P. Raczky, A. Richardson, P. Rowe, S. Rowland, I. Rowlandson, T. Saile, K. Sebők, W. Schier, G. Schmalfuß, S. Sharapova, H. Sharp, A. Sheridan, I. Shevnina, I. Sobkowiak-Tabaka, P. Stadler, H. Stäuble, A. Stobbe, D. Stojanovski, N. Tasić, I. van Wijk, I. Vostrovská, J. Vuković, S. Wolfram, A. Zeeb-Lanz and M.G. Thomas (2022). Dairying, diseases and the evolution of lactase persistence in Europe Nature. 608(7922): p. 336-345.
- Wilkin, S. (2022). The mystery of early milk consumption in Europe Nature. 608(7922): p. 268-269.
- Castro, J.P.L., M.N. Yancoskie, M. Marchini, S. Belohlavy, L. Hiramatsu, M. Kučka, W.H. Beluch, R. Naumann, I. Skuplik, J. Cobb, N.H. Barton, C. Rolian, and Y.F. Chan (2019). An integrative genomic analysis of the Longshanks selection experiment for longer limbs in mice [CC-BY]. eLife. 8: p. e42014. https://doi.org/10.7554/eLife.42014.

