17. Évolution du gout
Introduction au sujet
Dans ce chapitre, nous étudierons une composante du système sensoriel, le gout. Les récepteurs du gout permettent d’obtenir des informations cruciales sur l’environnement. C’est d’ailleurs une composante du système sensoriel qui a évoluée entre les espèces, résultant en des capacités différentes de percevoir leur environnement.
Objectifs spécifiques
À la fin de ce chapitre, vous serez en mesure de :
- Décrire les 5 gouts potentiellement perçus par les vertébrés et les récepteurs qui permettent de les percevoir
- Contraster le fonctionnement au niveau cellulaire des différents récepteurs du gout.
- Expliquer le lien entre la perception des différents types de gout et le fitness
- Caractériser l’origine évolutive de la diversité interespèce des récepteurs de l’amer
- Expliquer le processus de duplication de gènes et les différents destins possibles des gènes paralogues
- Comprendre les approches expérimentales utilisées pour tester l’évolution de la sensibilité d’un récepteur
- Expliquer le processus de reconstruction d’une séquence ancestrale
- Démontrer les mécanismes qui causent la variation interindividuelle de la sensibilité d’un récepteur de l’amer donné
- Expliquer le fonctionnement des récepteurs de l’umami et du sucré au niveau cellulaire
- Comprendre l’origine évolutive des récepteurs T1R
- Appliquer les concepts de pseudogénisation, de perte de gènes et de mutation à l’évolution du gout chez les vertébrés
- Comprendre le concept de l’écologie inversée : comment utiliser la séquence des génomes pour en apprendre sur le système sensoriel des espèces
Notions clés
- Le gout
- Les récepteurs cellulaires des cinq gouts
- Récepteur à canal ionique
- Récepteur couplé à une protéine G
- Lien entre la perception des cinq gouts et le fitness
- Les récepteurs T2R et l’amer
- Variation entre les espèces dans la perception de l’amer
- Les gènes paralogues et leur destin évolutif possible
- Variation du nombre de copies et dosage de gène
- Pseudogénisation et perte de gène
- Sous-fonctionnalisation
- Néo-fonctionnalisation
- Les arbres de gènes
- Écologie sensorielle
- Analyse fonctionnelle de séquences protéiques de différents récepteurs
- Reconstruction de la séquence ancestrale d’un récepteur et mutagenèse dirigée
- Variation entre les individus dans la perception de l’amer
- Le cas du récepteur T2R38 chez l’humain
- Combinaison des récepteurs de la famille T1R pour percevoir le sucré et l’umami
- Variation dans la perception du sucré entre différents groupes de vertébrés
- Un pseudogène pour un récepteur du gout chez les félins
- Perte d’un gène codant pour un récepteur du gout chez les oiseaux
- Mutation dans la séquence codante et évolution de la fonction d’un récepteur du gout chez les oiseaux
- Écologie inversée : traduire les informations sur le génome en information sur l’écologie
1. Le gout
La sous-section 1 est adaptée de [1].
La gustation est le sens particulier généralement associé à la langue chez l’humain. La surface de la langue, ainsi que le reste de la cavité buccale, est tapissée par un épithélium pavimenteux stratifié. Des bosses surélevées appelées papilles contiennent les structures de transduction gustative (figure 17.1). Dans la structure des papilles se trouvent des bourgeons gustatifs qui contiennent des cellules réceptrices gustatives spécialisées pour la transduction des stimuli gustatifs. Chez plusieurs espèces, des homologues des récepteurs du gout (des traits sont homologues lorsque leur similarité est due au partage d’un ancêtre commun) se retrouvent aussi sur la tête et sur le corps. On en retrouve aussi certains dans le système digestif et les reins.
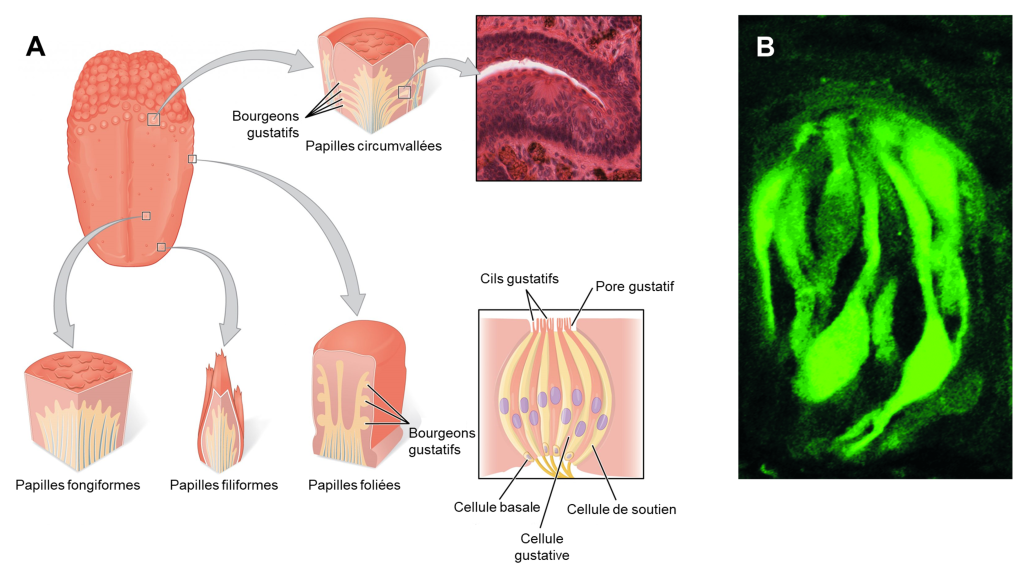
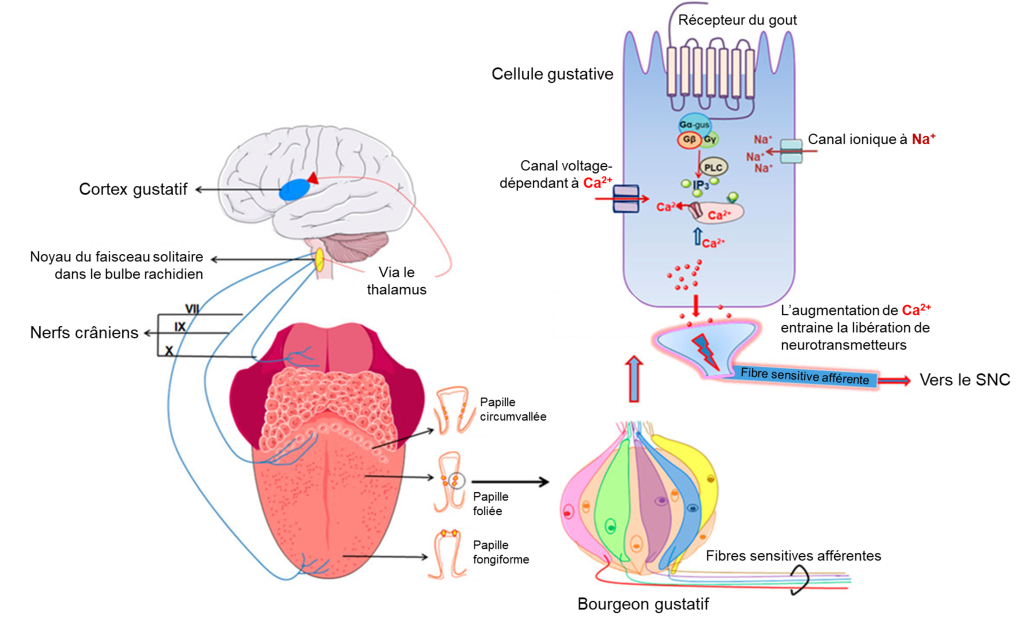
Une fois que les cellules gustatives sont activées par une molécule, elles libèrent des neurotransmetteurs sur les dendrites des neurones sensoriels. Chez l’humain, ces neurones font partie des nerfs crâniens facial et glossopharyngé, ainsi qu’un composant au sein du nerf vague dédié au réflexe nauséeux. La voie sensorielle de la gustation se déplace le long de ces nerfs, qui forment une synapse avec les neurones du noyau du faisceau solitaire du tronc cérébral (figure 17.2). Les axones du noyau du faisceau solitaire se projettent ensuite vers le noyau postérieur ventral du thalamus.
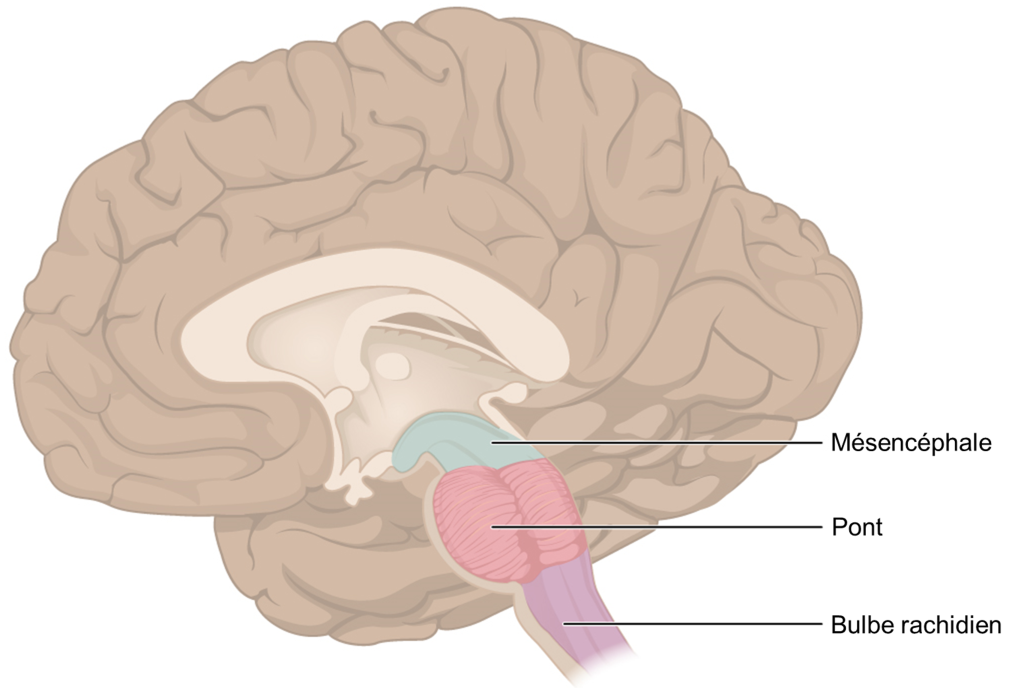
Enfin, les axones du noyau postérieur ventral se projettent vers le cortex gustatif du cortex cérébral, où le gout est traité et perçu consciemment comme une saveur (figure 17.3).
1.1 Les récepteurs cellulaires des cinq gouts
Cinq gouts principaux sont détectés par ces récepteurs chez différentes espèces de vertébrés : salé, acide/sûr, amer, sucré et umami. Des études récentes suggèrent que des récepteurs peuvent aussi détecter la présence de gras. Il est intéressant de noter que ce ne sont pas toutes les espèces de vertébrés qui perçoivent ces cinq gouts.
1.1.1 Récepteur à canal ionique
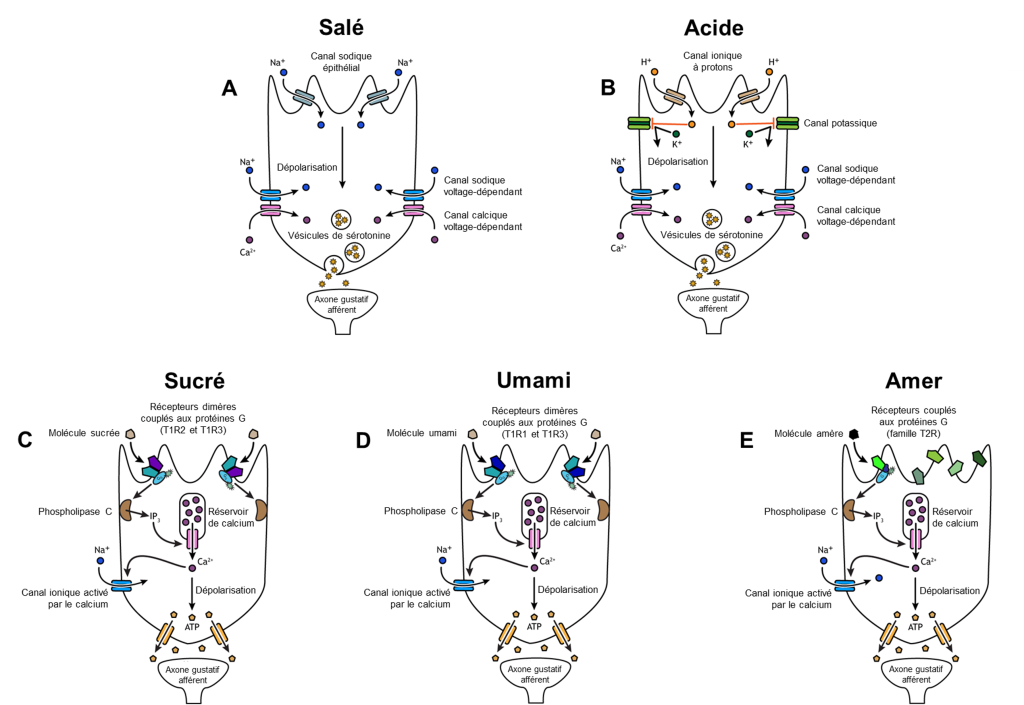
Si on veut comprendre l’évolution de la perception de ces gouts, on doit comprendre les mécanismes cellulaires qui mènent à leur perception. Deux gouts sont associés à des mécanismes simples. Le gout salé est simplement la perception des ions sodium (Na+) dans la salive. Lorsque vous mangez quelque chose de salé, les cristaux de sel se dissocient en ions Na+ et Cl–, qui se dissolvent dans la salive de votre bouche. La concentration de Na+ devient élevée à l’extérieur des cellules gustatives, créant un fort gradient de concentration qui entraine la diffusion des ions dans les cellules épithéliales (figure 17.4A). Le sodium passe la membrane par un canal ionique qui est sélectivement perméable au sodium. C’est le même type de canal qui est exprimé dans nos néphrons qui jouent un rôle dans la réabsorption d’ions dans le rein. L’entrée de Na+ dans ces cellules entraine la dépolarisation de la membrane cellulaire, ce qui mène au relâchement de neurotransmetteurs dans la fente synaptique et l’activation d’un neurone afférent du système sensoriel. Le gout acide est la perception de la concentration en H+. Tout comme les ions sodium dans les saveurs salées, ces ions hydrogène provenant de nourriture acide pénètrent dans la cellule et déclenchent sa dépolarisation (figure 17.4B). La dépolarisation de la membrane cellulaire mène aussi au relâchement de neurotransmetteur dans la fente synaptique, ce qui envoie un signal au neurone sensoriel afférent. Les scientifiques dans ce domaine se sont longtemps demandé comment une si large gamme de pH pouvait être détectée par un seul type de récepteur. Des expériences suggèrent que chez les mammifères, des récepteurs contenus dans les papilles gustatives expriment un canal ionique de la famille des TRP qui permet aussi de détecter l’acide. De plus, un canal ionique sélectif aux protons qui détecte les ions H+ à de faibles niveaux d’acidité a aussi été découvert récemment, l’otopétrine [6]. Ainsi, différents récepteurs détectent différents niveaux d’acidité. Ces découvertes nous permettent de mieux comprendre les mécanismes impliqués dans ce gout.
1.1.2 Récepteur couplé à une protéine G
Les gouts sucrés, umamis et amers résultent de molécules alimentaires se liant à un récepteur couplé à une protéine G (un « GPCR »). Un système de transduction du signal de la protéine G conduit éventuellement à la dépolarisation de la cellule gustative.
Il y a plusieurs différences entre ces récepteurs : les récepteurs du sucré et de l’umami sont formés de deux protéines distinctes qui forment un hétérodimère alors que les récepteurs de l’amer sont composés d’une seule protéine ancrée dans la membrane cellulaire des cellules gustatives (figure 17.4 C, D et E). De plus, les gènes qui codent pour les récepteurs du sucré et de l’umami sont conservés évolutivement lorsqu’on compare leur séquence entre différents groupes de vertébrés, alors que les gènes qui codent pour les récepteurs de l’amer varient en nombre dans le génome d’une espèce donnée et en séquence pour un gène orthologue entre les espèces [12].
Le gout sucré est la sensibilité des cellules gustatives à la présence de glucose dissout dans la salive. D’autres monosaccharides tels que le fructose ou des édulcorants artificiels tels que l’aspartame, la saccharine ou le sucralose activent également les récepteurs sucrés (figure 17.4C). L’affinité de ces récepteurs pour chacune de ces molécules varie, et certaines auront un gout plus sucré que le glucose, car elles se lient différemment au récepteur couplé aux protéines G.
Le gout umami a pour origine le mot « délicieux » en japonais. Comme le sucré, il repose sur l’activation des récepteurs couplés aux protéines G par une molécule (figure 17.4 D). La molécule qui active ce récepteur est l’acide aminé L-glutamate. Par conséquent, la saveur umami est souvent perçue en mangeant des aliments riches en protéines. Sans surprise, les plats qui contiennent de la viande sont souvent décrits comme umami. La saveur umami est aussi très présente dans les champignons, la sauce soya, le miso, la sauce Worcestershire et la sauce de poisson (Nước mắm), ainsi que dans la levure alimentaire et certains fromages comme le parmigiano. C’est le glutamate contenu dans ces produits que l’on perçoit avec nos récepteurs umami. Le ketchup est un autre exemple d’aliment umami. La prochaine fois qu’on vous fait un commentaire lorsque vous inondez votre repas de ketchup, répondez que c’est umami!
Le gout amer est similaire au sucré et à l’umami en ce sens que les molécules alimentaires se lient à des récepteurs couplés à une protéine G (figure 17.4E). Cependant, cela peut se produire de différentes manières, car il existe une grande diversité de molécules au gout amer. Un groupe majeur de molécules au gout amer sont les alcaloïdes. Les alcaloïdes sont des molécules contenant de l’azote que l’on trouve couramment dans les produits végétaux au gout amer, tels que le café, le houblon (dans la bière), les tanins (dans le vin), le thé et l’aspirine. Puisque les alcaloïdes sont souvent toxiques, les plantes qui contiennent des alcaloïdes sont ingérées moins souvent et/ou en moins grande quantité par les herbivores. D’autres plantes produisent du glucosinolate, un composé qui est aussi perçu comme amer et qui a une fonction de défense face aux herbivores chez les plantes crucifères, tel que le brocoli ou le chou. Il a récemment été démontré que certains récepteurs de l’amer peuvent détecter la présence de bactéries lorsqu’ils sont exprimés dans des cellules épithéliales des voies respiratoires. Ils seraient ainsi associés à la défense immunitaire en plus de détecter l’amer [13].
Exercices
Les récepteurs du gout
1.2 Lien entre la perception des cinq gouts et le fitness
La détection de différentes molécules présentes dans l’environnement a vraisemblablement une influence sur le fitness. On retrouve déjà la capacité de détecter des molécules qui vont affecter l’organisme chez les unicellulaires, la chimiomotaxie, où par exemple un eucaryote unicellulaire va réagir à une molécule détectée dans l’espace en se dirigeant vers la source ou en s’en éloignant. Chez les êtres multicellulaires, plusieurs animaux recherchent des aliments comme les fruits, qui ont un gout sucré. Ces aliments sont une source précieuse de glucide et donc d’énergie et la valeur adaptative d’avoir la capacité de détecter de tels aliments est probablement élevée (figure 17.5A). Les récepteurs du gout amer jouent un rôle important en permettant d’éviter l’ingestion de substances toxiques (telles que les alcaloïdes végétaux) chez les omnivores. Par exemple, certains composés amers inhibent l’absorption d’iode chez les humains, ce qui cause des problèmes de synthèse de l’hormone thyroïdienne. Il y a donc possiblement un avantage à détecter les alcaloïdes nuisibles à la santé. En effet, les humains mangent de nombreux produits végétaux qui contiennent ces alcaloïdes, par exemple le cacao, la quinine, le café ou le thé (figure 17.5B). Finalement, le gout umami indique la présence de protéine, un autre apport nutritionnel vital.
Dans les prochaines sections, nous verrons comment fonctionnent les récepteurs de l’amer, du sucré et de l’umami au niveau cellulaire ainsi que leur évolution.

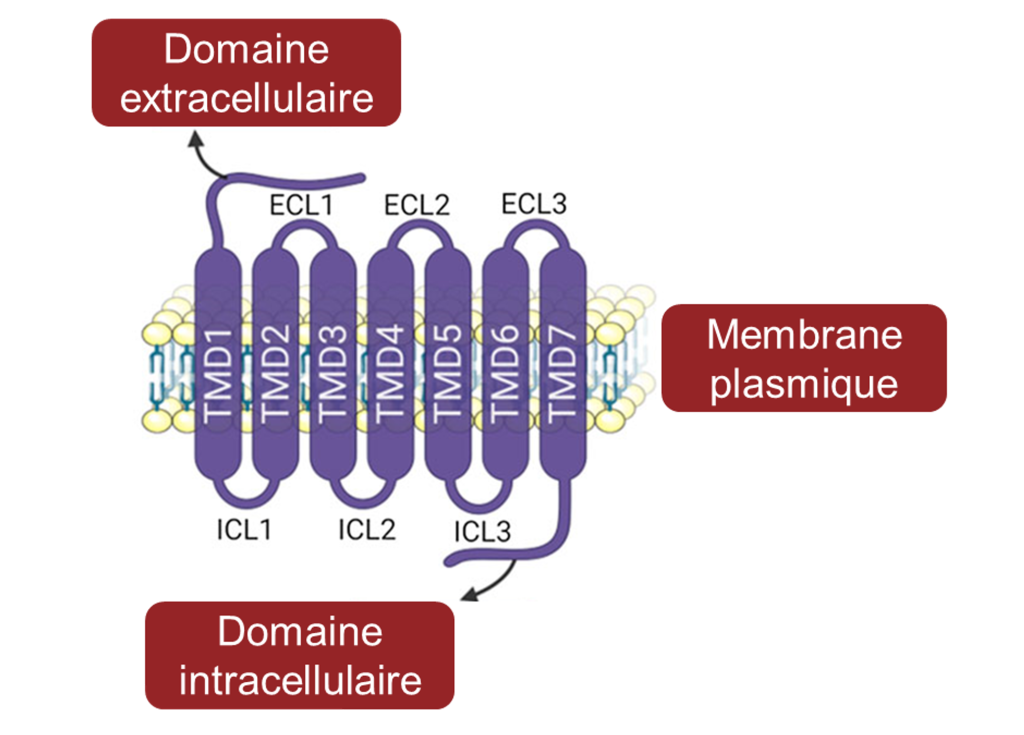
2. Les récepteurs T2R et l’amer
Nous allons d’abord aborder le cas des récepteurs de l’amer qui sont dans une classe à part comparativement aux récepteurs du sucré et de l’umami. Ce récepteur est composé d’une seule protéine de type T2R ancrée dans la membrane cellulaire des cellules gustatives (figure 17.6). Le nombre de récepteurs de l’amer a évolué entre les espèces, ce qui résulte en des récepteurs de la famille de gène T2R avec des séquences différentes qui perçoivent présumément différentes molécules amères et un nombre total de gènes codant pour un récepteur de l’amer qui diffère entre les espèces. Nous allons maintenant voir pourquoi le nombre de récepteurs de l’amer varie entre des groupes de vertébrés et comment on peut mesurer que leur sensibilité à des composés amers a évolué de façon divergente. Nous nous attarderons ensuite à la variation intra-espèce en étudiant un des récepteurs chez l’humain et son association avec la variation de sa sensibilité entre les individus pour une molécule amère particulière, le glucosinolate.
2.1 Variation entre les espèces dans la perception de l’amer
Les récepteurs T2R sont apparus chez les poissons cartilagineux. Il y a plusieurs récepteurs de l’amer dans le génome chez les vertébrés. Les biologistes évolutifs qui s’intéressent aux variations dans le génome de différentes espèces ont démontré qu’il y a 25 gènes codant pour des protéines T2R chez les humains, tandis que les rats en ont 35, les chiens 16, les ours polaires 13 et les chats 12. La moyenne du nombre de récepteurs de l’amer chez les amphibiens qui ont été étudiés jusqu’à maintenant est de 93 et une espèce en a même 186 (figure 17.7). Les chercheurs et chercheuses qui ont étudié ces génomes d’amphibiens ont mesuré le moment où ces récepteurs sont exprimés et ont découvert que les récepteurs exprimés au stade têtard ne sont pas les mêmes que ceux qui sont exprimés au stade adulte [19, 20].
L’arbre phylogénétique de la figure 17.7 nous montre que les récepteurs de l’amer sont apparus chez les poissons osseux et que les poissons cartilagineux (requins, raies, chimères) ne perçoivent pas les molécules amères, mais une étude très récente semble suggérer que l’ancêtre des requins et des poissons osseux avait déjà un récepteur de l’amer dans leur génome et que ceux-ci sont apparus chez les animaux à mâchoire [21]. Les biologistes évolutifs ont déterminé que c’est la perception du gout qui a évolué le plus récemment des 5 gouts possiblement détectés par les vertébrés.
![Texte alternatif : À gauche, on trouve un cladogramme de plusieurs groupes de vertébrés avec un nombre sur la branche qui représente le nombre d’espèces étudiée par groupe (entre parenthèses ci-après) : les mammifères (52), les oiseaux (48), les reptiles (19), les amphibiens (22), les poissons à nageoires charnues (1), les poissons à nageoires rayonnées (23) et les poissons cartilagineux (1). À droite, vis-à-vis chaque branche de l’arbre, on retrouve une boite à moustache, qui représente le nombre de gènes Tas2r intact dans chaque groupe. Les valeurs minimales, moyennes et maximales sont également indiquées entre crochets, comme suit : mammifères [0, 13.8, 36], oiseaux [0, 2.4, 7], reptiles [0, 8.1, 50], amphibiens [22, 93.8, 186], poisson à nageoires charnues [74], poissons à nageoires rayonnées [0, 4.8, 21], poissons cartilagineux [0].](https://pressbooks.etsmtl.ca/app/uploads/sites/5/2024/01/U3C17F7-1024x422.png)
2.1.1 Les gènes paralogues et leur destin évolutif possible
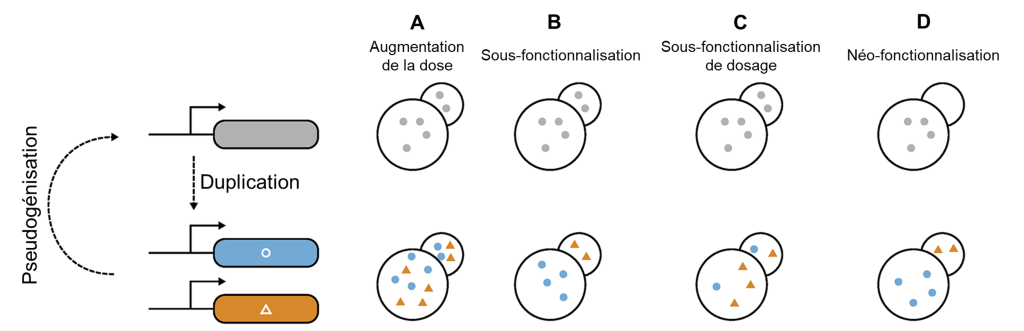
Ces différentes protéines qui détectent l’amer sont le résultat d’un processus de duplication suivi de divergence (Figure 17.8). Nous avons vu brièvement le processus de duplication de gène lorsque nous avons vu les types de variation génétique qui existent (chapitre 2). Lorsqu’il y a duplication, il y a 2 copies du gène dans le génome. Ces deux copies s’appellent des paralogues. Dans le cas de la famille des protéines T2R, il y a eu plusieurs duplications successives. On a donc ce qu’on appelle une « famille » de gènes.
2.1.2 Variation du nombre de copies et dosage de gène
Quand un gène est dupliqué, le premier effet est que le dosage est habituellement doublé (pas toujours, mais par simplicité on aura comme prémisse que c’est ce qui se passe dans la majorité des cas). Si on avait 6 ARN messagers synthétisés, on en a maintenant 12 et donc on double aussi le nombre de protéines traduites. Avoir le double de gène, ou le quadruple, etc., permet de synthétiser une plus grande quantité de la protéine en question. Le « dosage du gène » (« gene dosage ») est relié à la variation du nombre de copies. La protéine n’a pas été modifiée, il n’y a pas de mutation dans la séquence codante ou même la séquence régulatrice, c’est uniquement le nombre de copies qui change (figure 17.8A). Ceci pourrait être un avantage dans certains cas précis, par exemple si le gène code pour une protéine qui est une enzyme et qu’avoir plus de cette enzyme augmente la performance au niveau cellulaire, ce qui à son tour améliore le fitness de l’organisme. Cependant, il peut aussi être très néfaste d’avoir un plus grand nombre de copies, quand l’augmentation de la quantité d’une protéine en particulier peut avoir des effets négatifs pour la cellule. Par exemple, ça peut être le cas lorsque d’autres protéines qui interagissent avec celle qui est dupliquée n’ont pas été dupliquées et n’ont donc pas doublé en quantité. Dans ce cas, la stœchiométrie de la cellule se trouve débalancée. On le voit dans certaines maladies humaines qui sont liées à une augmentation du nombre de copies. Dans ce cas-là, un individu avec cette duplication aura un fitness moindre et la duplication disparaitra au fil des générations.
2.1.3 Pseudogénisation et perte de gène
Le destin le plus courant de la deuxième copie est qu’elle ne se fixe pas dans la population et disparait (figure 17.8). L’une des deux copies de gènes perd sa fonction en accumulant des mutations au cours des générations et elle disparait. La perte du paralogue s’appelle la pseudogénisation si le gène est encore dans le génome, mais est non fonctionnel à cause des mutations. Éventuellement, le pseudogène sera perdu, car il n’y a plus de sélection pour sa fonction. Dans le cas où la copie du gène est carrément disparue du génome, on parle de perte de gène. On revient alors à une seule copie du gène.
2.1.4 Sous-fonctionnalisation
Une autre possibilité de destin pour ces paralogues est la sous-fonctionnalisation, c’est-à-dire que l’évolution mène à une division de la fonction totale du gène ancestral en deux fonctions uniques, mais apparentées (figure 17.8B). Une fois qu’il y a deux copies, des mutations peuvent se produire dans une copie et non dans l’autre, ce qui fait qu’ils évoluent indépendamment, comme dans le cas de la pseudogénisation et la perte de gène. Cependant dans ce cas-ci, les deux copies sont conservées et demeurent fonctionnelles. La mutation peut être dans la séquence codante, ce qui peut affecter la séquence d’acides aminés qui forme cette protéine. La mutation pourrait aussi être dans la séquence régulatrice et changer l’expression de la protéine (son niveau, l’endroit où elle est exprimée, le moment où elle est exprimée).
2.1.5 Néo-fonctionnalisation
Le troisième destin possible pour les paralogues est que l’une des deux copies du gène acquière une nouvelle fonction à la suite d’une ou de plusieurs mutations. C’est la néo-fonctionnalisation (figure 17.8C). C’est ce destin qui nous intéresse dans le cas des récepteurs de l’amer. En effet, différents gènes codant pour des récepteurs de l’amer détectent des molécules différentes. Certaines espèce sont moins de types de récepteurs de l’amer par perte de gène plus tard durant l’évolution.
Aubin-Horth, N. (2024). Écophysiologie évolutive: les gènes paralogues et leur destin évolutif. [Vidéo]. Youtube. CC-BY. https://youtu.be/FWJInQwXwbo?si=4rlFWuDk7ymmEBtu
Vous trouverez la transcription de l’audio de cette capsule en cliquant sur ce lien: Transcription_format_word
Exercices
Destin évolutif des paralogues
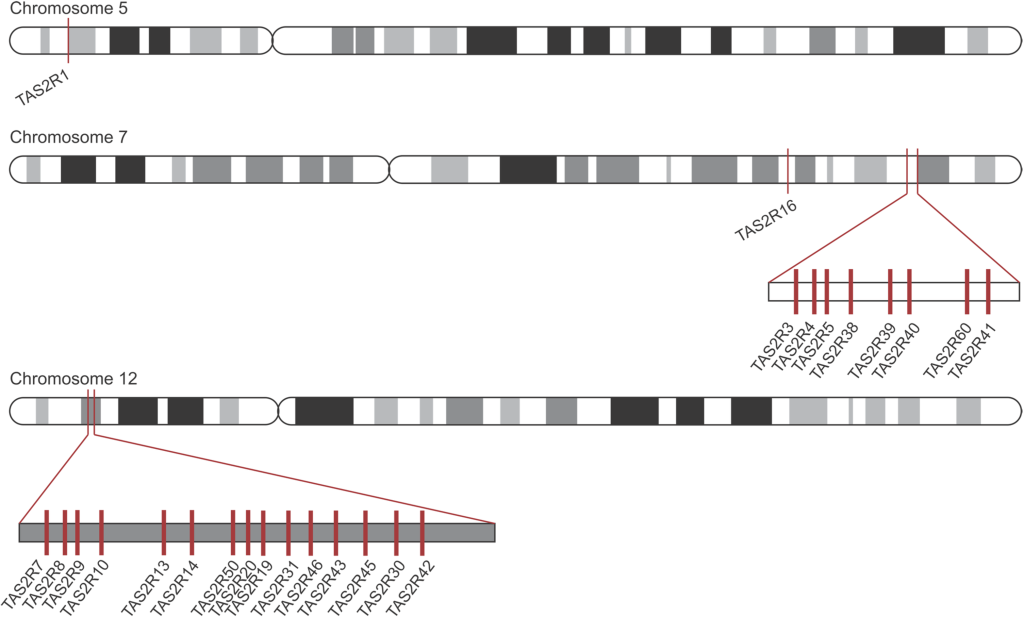
On peut utiliser l’exemple du génome des humains pour mieux comprendre le rôle de la duplication de gènes dans l’évolution des récepteurs de l’amer, car on a une idée précise de la localisation des paralogues sur les chromosomes (figure 17.9). Le fait que les gènes aient une forte similarité de séquence et qu’ils sont près physiquement l’un de l’autre sont des indices que ce sont des paralogues qui ont évolué par duplication. Chaque gène de cette famille de récepteur code pour un récepteur T2R différent. Les biologistes évolutifs ont émis l’hypothèse que ces différents récepteurs permettent de détecter une molécule amère différente, et donc que la fonction des paralogues aurait évoluée, ce qui serait un exemple de néo-fonctionnalisation. Il faut cependant souligner que pour plusieurs récepteurs, on assume que le changement de séquence affecte sa sensibilité et le type de molécule qui est reconnu, mais ceci n’a pas toujours été testé directement. Il y a donc encore du travail à faire par les physiologistes pour découvrir l’effet des divergences de séquence entre les paralogues sur leur capacité à détecter différentes molécules.
2.2 Les arbres de gène
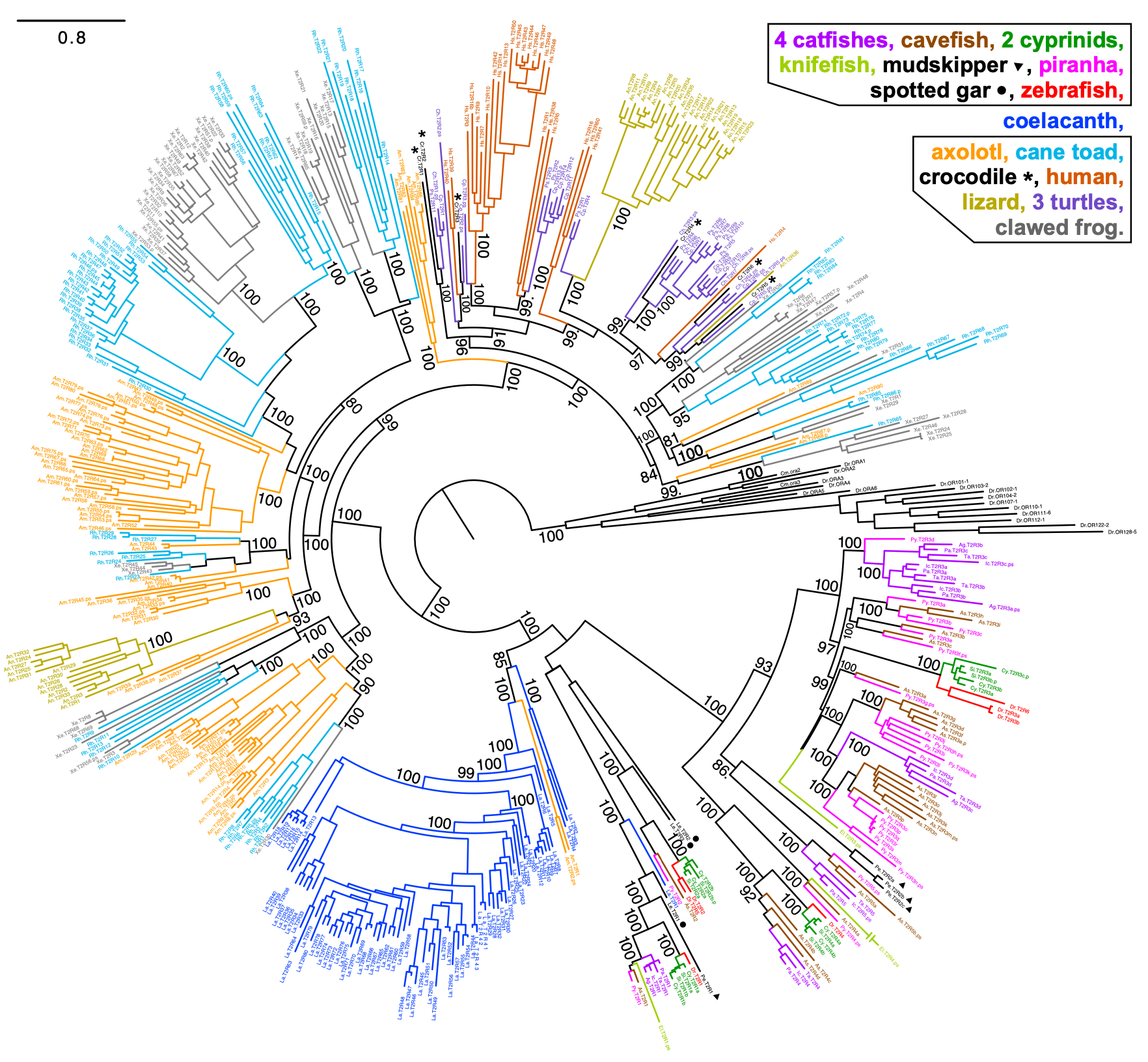
Si on regarde l’évolution de la séquence des gènes qui codent pour les récepteurs de l’amer chez différents vertébrés, on peut voir qu’ils ont évolué de façon indépendante à de nombreuses reprises (figure 17. 20). Comme pour l’exemple des humains, chaque paralogue a un numéro qui l’identifie (« T2R4 », « T2R8 »). On peut faire un arbre phylogénétique en utilisant uniquement les séquences de gènes qui codent pour des récepteurs de l’amer dans différentes espèces. Selon le patron que prend ce type spécifique d’arbre phylogénétique, ceci nous indique comment la séquence a évolué, c’est à dire si elle a évolué une seule fois dans un ancêtre commun des différentes espèces et a été conservée dans la plupart ou la totalité d’entre elles ou si la séquence a évoluée de façon indépendante. Dans le cas des récepteurs de l’amer, les séquences se ressemblent plus entre elles à l’intérieur d’une espèce (représenté par une couleur sur la figure 17.20) et ceci suggère une évolution indépendante par duplication et perte de gènes qui donne le nombre variable de récepteurs de l’amer selon les groupes. Si les séquences avaient évolué une seule fois par duplication puis avaient été conservées, les séquences d’un gène donné pour plusieurs espèces auraient été regroupées sur une branche (ce que nous verrons plus loin avec les gènes codant pour les récepteurs du sucré et de l’umami).
Les biologistes qui étudient l’évolution du système sensoriel se sont demandé quel pourrait être l’agent de sélection qui favoriserait la conservation des paralogues qui ont une nouvelle fonction. Une étude a démontré que chez les tétrapodes (les amphibiens, les reptiles, les oiseaux et les mammifères), il y a une corrélation entre la taille du répertoire de gènes codants pour un récepteur de l’amer et la proportion de plantes ou d’insectes dans la diète. Plus la diète en contient, plus l’espèce a un grand nombre de récepteurs différents. Ces sources de nourriture contiennent souvent des toxines et l’hypothèse est que les différents récepteurs T2R sont sensibles à des composés amers différents et donc que les espèces qui se nourrissent de plusieurs espèces de plantes et d’insectes ont subi une pression de sélection pour une meilleure capacité à détecter les toxines de leur diète [24]. Cette étude montre une corrélation entre les deux variables. Nous verrons plus loin comment on peut tester expérimentalement que les récepteurs de l’amer avec des séquences différentes ont effectivement des sensibilités à différentes molécules amères.
2.3 L’écologie sensorielle
Comme les humains et d’autres mammifères, les singes ont aussi plusieurs gènes de la famille des récepteurs de l’amer dans leur génome qui codent pour des récepteurs différents. Des biologistes travaillant dans le domaine de « l’écologie sensorielle » se sont intéressés à la relation entre l’écologie des espèces de singes du Nouveau Monde et l’évolution de leur système sensoriel. Leur hypothèse de départ était qu’on devrait voir une association entre les deux. Les espèces de singe choisies pour l’étude sont intéressantes, car elles ont des sources de nourriture différentes : fruits, noix, feuilles, fleurs, exsudats de plante, insectes. De plus, ces singes vivent dans des niches temporelles différentes (figure 17.11). Par exemple, le Douroucouli (owl monkey) est 100 % nocturne et est frugivore, avec aussi quelques insectes, des fleurs, du nectar et des feuilles dans sa diète (figure 17.11A). En comparaison, les ouistitis (un type de marmoset) sont diurnes et certaines espèces se nourrissent de la gomme des arbres et d’insectes (figure 17.11B).

2.3.1 Analyse fonctionnelle de séquences protéiques de différents récepteurs
Les biologistes ont étudié deux gènes codant pour deux récepteurs de l’amer: T2R1 et T2R4. Les séquences de ces deux gènes diffèrent, c’est pourquoi ils ont un nom formé de « T2R » suivi d’un chiffre qui les identifie comme des paralogues membres de la même famille de gènes. De plus, la séquence diffère entre les espèces de singes pour un gène donné, par exemple la séquence de T2R1 n’est pas exactement la même pour les douroucoulis et les ouistitis. On suppose donc que les singes ont des capacités de détection de molécules amères différentes, mais quelles sont ces molécules? Lorsqu’on étudie les humains, on peut leur demander si un composé en particulier goute amer ou non, mais avec d’autres espèces, on utilise plutôt l’activité du récepteur en réponse à un composé, que l’on peut mesurer en laboratoire. Un récepteur activé envoie habituellement un signal d’amertume au cerveau. On peut donc mesurer l’activation de la cellule portant le récepteur de l’amer directement dans un pétri et savoir ce que le cerveau recevrait comme signal! Les biologistes se sont ainsi servis d’une analyse fonctionnelle dans une lignée cellulaire pour quantifier l’activité des récepteurs de chaque espèce en réponse à différents composés amers, sans faire d’expérience sur des animaux. Ils ont simplement eu à récolter de l’ADN de chaque espèce pour séquencer les gènes qui les intéressaient (ce qu’ils ont fait avec de la salive et des fèces) afin de pouvoir construire une lignée cellulaire pour chaque séquence de récepteur. D’ailleurs, les physiologistes qui étudient l’évolution du gout amer chez les humains et qui veulent tester des centaines de molécules pour déterminer lesquelles sont détectées par quelle copie du gène TAS2R et à quelle concentration utilisent aussi des lignées cellulaires plutôt que des sujets humains [5].
Lors de l’activation du récepteur T2R par des ligands amers, une cascade de signalisation conduit à la libération de Ca2+, entrainant un afflux de Na+ via des canaux (voir figure 17.4E). Cet afflux de Na+ dépolarise la membrane cellulaire et envoie un signal au neurone sensoriel associé à la cellule gustative. On peut donc mesurer ce flux de calcium dans une cellule en laboratoire et ainsi estimer quel aurait été le signal envoyé au système nerveux. Si l’on créé une lignée cellulaire avec la version de notre récepteur d’intérêt, disons la séquence du récepteur T2R1 du ouistiti, on peut faire un test de réponse du récepteur avec différentes concentrations de substances variées connues pour leur amertume (ici la colchicine et le camphre) et mesurer l’augmentation (ou non) du flux de calcium. On peut ainsi faire un portrait de la sensibilité des différents singes basé sur cette réponse cellulaire. Avec cette méthode, les physiologistes ont découvert que les douroucoulis ont un récepteur T2R1 très sensibles au camphre (figure 17.12A) et que les ouistitis ont un récepteur T2R4 sensible à la colchicine (figure 17.12B).
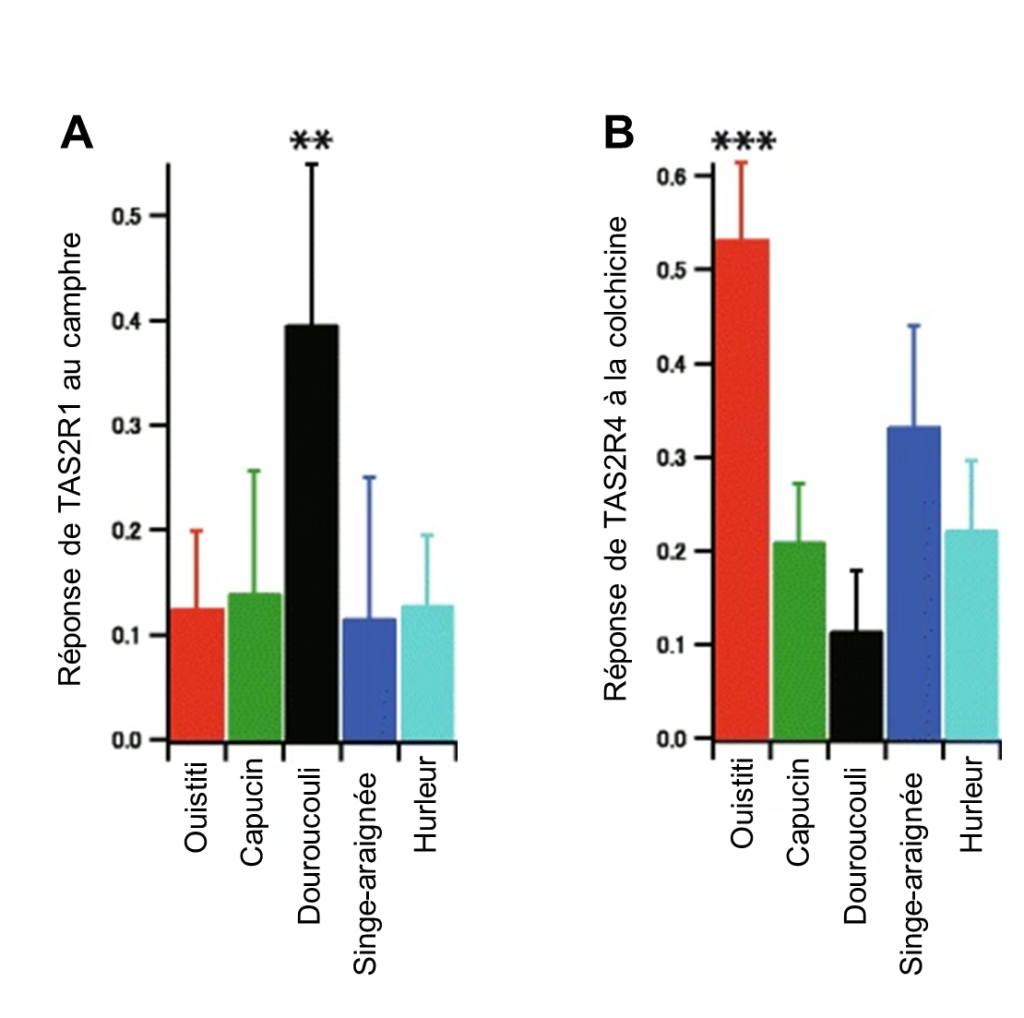
2.3.2 Reconstruction de la séquence ancestrale d’un récepteur et mutagenèse dirigée
Les biologistes ont ensuite combiné cette approche à une analyse des séquences de chaque récepteur qui leur a permis de « reconstruire » la séquence du récepteur ancestral en déduisant la séquence la plus probable. Cette reconstruction leur a donné la séquence en acides aminés probable du récepteur qui est l’ancêtre des récepteurs présents maintenant. Ils avaient la séquence de la protéine, mais pas d’information sur son activité. Comme dans la première expérience, ils ont inséré ces récepteurs ancestraux dans une lignée cellulaire et ont pu tester leur sensibilité à l’amer, comme avec une machine à voyager dans le temps! Ils ont ainsi pu déterminer à quel moment durant l’évolution la sensibilité à ces molécules a changé pour chaque espèce.
De plus, comme ils savaient quelles mutations ont eu lieu durant l’évolution lorsque les espèces de singe ont divergé, ils ont pu déduire quelle mutation était responsable du changement de sensibilité à l’amer parmi les nombreuses mutations dans la séquence du gène. En effet, ce ne sont pas toutes les mutations qui affectent l’acide aminé produit et même si l’acide aminé est modifié, ça n’a pas toujours un effet sur la fonction de la protéine. Ils ont prédit quel changement d’acide aminé semblait être lié à un changement de sensibilité à l’amer durant l’évolution de ces singes. Ils ont ensuite utilisé la mutagenèse dirigée afin de créer un récepteur avec cette mutation uniquement, ce qui leur a permis de mesurer l’effet de cette mutation sur la sensibilité du récepteur, confirmant leur prédiction. Tout ça sans toucher à un seul singe! Les molécules amères utilisées dans l’expérience ont été choisies parce que ce sont des molécules détectées par les humains. Il reste donc à faire le lien entre la sensibilité de chaque espèce et les molécules amères présentes dans leur diète qui diffère, afin de véritablement répondre à la question d’écologie sensorielle, c’est-à-dire : « est-ce qu’il y a une relation entre l’écologie des espèces de singes du Nouveau Monde et l’évolution de leur système sensoriel? »
2.4. Variation entre les individus dans la perception de l’amer
Si on se base sur le nombre de récepteurs différents, on peut émettre l’hypothèse que les humains ont d’excellentes capacités à percevoir l’amer, comparé à d’autres espèces de mammifères, comme le chien. Cependant, il y a aussi des variations génétiques à l’intérieur des humains. Pensons au gout du brocoli, qui est aimé par certains et détesté par d’autres, et ce avec une haute héritabilité (figure 17.13). Vous avez peut-être même quelqu’un en tête en lisant ces lignes! On peut proposer deux mécanismes par lesquels ces différences de phénotype, où certaines personnes goutent fortement l’amer et d’autres non. La première prédiction est qu’il y a des différences dans la séquence codante de la protéine du récepteur T2R qui détecte cette molécule, c’est-à-dire qu’il y a une mutation qui change la fonction de la protéine chez certaines personnes, ce qui affecte le fonctionnement du récepteur, possiblement en modifiant la capacité de lier une molécule amère. La deuxième prédiction est qu’il y a des différences dans la séquence régulatrice, c’est-à-dire une mutation qui change le niveau d’expression du gène qui code pour le récepteur T2R et qui change donc la quantité de celui-ci entre les personnes. Évidemment, les deux prédictions peuvent être vraies et on peut à la fois avoir une mutation qui affecte la sensibilité du récepteur et une autre mutation qui affecte sa quantité.

2.4.1 Le cas du récepteur T2R38 chez l’humain
On sait depuis longtemps qu’il y a des différences interindividuelles dans la perception de l’amer chez l’humain. De nombreuses expériences ont été faites en donnant un composé amer, le PTC (phénylthiocarbamide) et le PROP (propylthiouracile) à des personnes et en leur demandant de déterminer l’amertume de ces produits. Dans plusieurs études menées chez des populations d’ascendance européenne, les gens se classent en deux groupes : ceux et celles qui goutent l’amer et ceux et celles qui ne perçoivent pas l’amertume de ces deux composés. Des biologistes ont découvert qu’un des gènes qui code pour un récepteur T2R, le TAS2R38 existe en plusieurs allèles, c’est-à-dire qu’il y a des variations dans la séquence d’ADN à ce locus entre les individus, spécifiquement dans la séquence codante. Ces substitutions de nucléotides sont non synonymes (ce sont des mutations faux-sens), ce qui veut dire que le changement de codon causé par le changement de nucléotides entraine la traduction d’un acide aminé différent. À ce jour, c’est le seul gène des 25 gènes de la famille des T2R qui est connu pour avoir plusieurs allèles chez l’humain. On nomme les allèles de ce gène en se basant sur les acides aminés qui sont codés différemment selon le polymorphisme de nucléotides aux positions 49, 262 et 296 [30]. Les deux allèles retrouvés couramment en Amérique du Nord, en Europe et en Asie sont l’allèle PAV (Proline, Alanine, Valine) et AVI (Alanine, Valine, Isoleucine). Il existe d’autres allèles qui ont une fréquence plus faible en Amérique du Nord, en Europe et en Asie (< 5 %) (AAV et AAI) que l’on retrouve cependant à plus haute fréquence en Afrique (figure 17.14). Les biologistes ont voulu vérifier si ces différences d’allèles (PAV et AVI) sont associées aux différences de capacité de gouter l’amer déjà mesurées chez les humains. En d’autres mots, est-ce que le fait d’avoir un allèle ou l’autre affecte directement le gout amer perçu par ces récepteurs [31]? Ils se sont spécifiquement intéressés aux plantes qui contiennent du glucosinolate, comme les crucifères (chou, brocoli, moutarde).
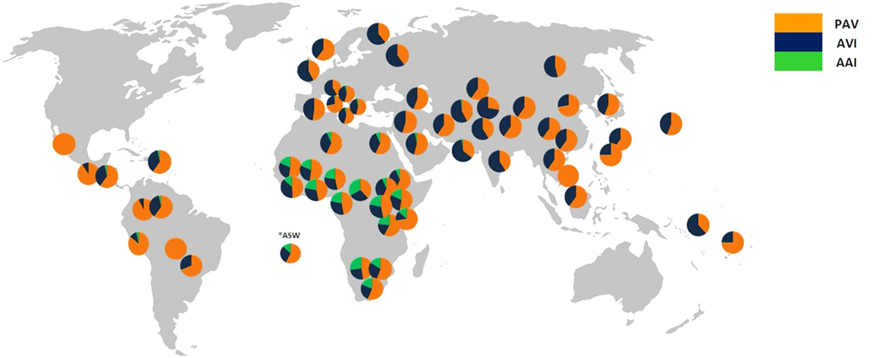
Les scientifiques ont fait gouter différents légumes reconnus comme contenant du glucosinolate ou non à des personnes dont on connaissait le génotype pour le gène TAS2R38 (figure 17.15). Les personnes étaient homozygotes PAV, homozygotes AVI ou hétérozygotes PAV/AVI (c’est-à-dire qu’ils avaient un allèle de chaque type). Chaque personne donnait un score d’amertume à l’échantillon, après s’être entrainée avec de la quinine, le composé que l’on retrouve dans le tonique pour faire des « gin & tonic » et qui représente une amertume moyenne.

Les biologistes ont déterminé que la présence d’un allèle ou l’autre au gène TAS2R38 est en effet associée avec une perception différente de l’amer pour le même aliment. Les individus qui ont le génotype PAV/PAV goutent beaucoup l’amer et les individus qui ont le génotype AVI/AVI ne perçoivent pas l’amertume. De façon intéressante, les personnes hétérozygotes PAV/AVI donnent un score intermédiaire d’amertume aux aliments [31]. Cette différence marquée était vraie pour les aliments contenant du glucosinolate seulement. En effet, les autres légumes sans glucosinolate (des endives, du radicchio, de l’aubergine…) ont reçu un score d’amertume similaire, peu importe l’allèle présent au gène TAS2R38. L’allèle que vous avez à ce gène affecte donc votre capacité à gouter l’amertume du brocoli parce qu’il contient du glucosinolate et c’est cette molécule qui active le récepteur.
3. Combinaison des récepteurs de la famille T1R pour percevoir le sucré et l’umami
Les gènes qui codent pour les récepteurs du sucré et de l’umami sont apparus durant l’évolution chez l’ancêtre des poissons cartilagineux et forment la famille de gènes T1R. Contrairement au récepteur de l’amer, les récepteurs du sucré et de l’umami sont composés de deux protéines qui forment des hétérodimères (figure 17.16). Lorsque la protéine T1R3 forme un dimère avec la protéine T1R2, la structure détecte le sucré. Lorsque T1R3 forme un dimère avec T1R1, le récepteur résultant détecte l’umami. C’est pourquoi il y a un total de trois gènes (T1R1, T1R2 et T1R3) qui sont nécessaires pour avoir la perception de deux gouts (sucré et umami). En effet, T1R1 et T1R2 ne sont pas fonctionnels s’ils ne sont pas en dimère avec T1R3.
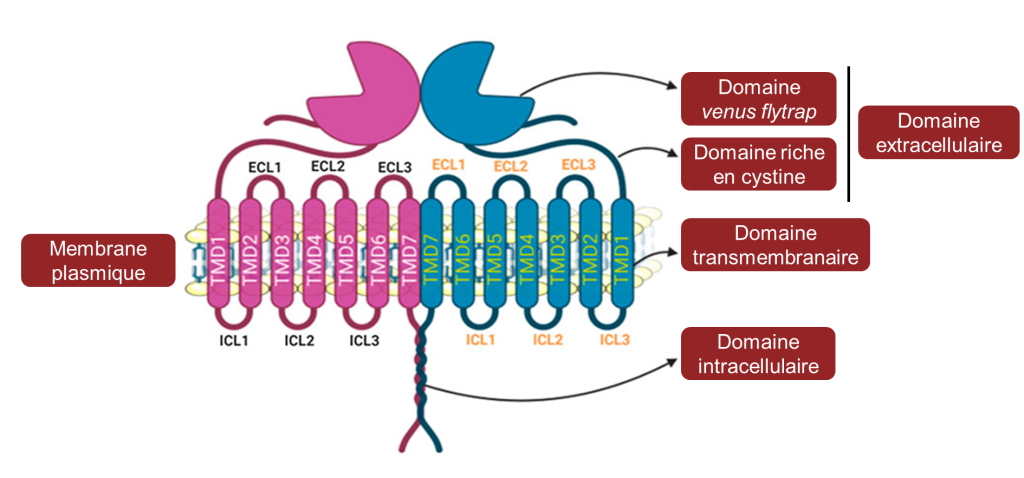
3.1 Variation dans la perception du sucré entre différents groupes de vertébrés
L’analyse phylogénétique des gènes T1R disponibles pour les espèces de vertébrés a montré que les gènes T1R1, T1R2 et T1R3 ont évolué par duplication avant la divergence évolutive des poissons et des tétrapodes. Les récepteurs sensibles au sucré et au gout umami sont remarquablement conservés — c’est-à-dire que leur nombre et leur séquence n’ont pas beaucoup changé au cours des générations et reste similaires entre les groupes de vertébrés. Ainsi, chez les tétrapodes, on ne trouve que les gènes T1R1, T1R2 et T1R3 (il existe d’autres gènes de cette famille chez certains groupes de poissons mais leur fonction est mal connue). Ceci contraste par exemple avec les gènes qui codent pour les récepteurs de l’amer qui sont en nombre varié entre les espèces de vertébré.
Certains groupes de vertébrés ont perdu la capacité de percevoir le sucré. Par exemple, les félins ont tous perdu la capacité de détecter le sucré, dû à une perte de fonctionnalité du gène Tas1r2 qui code pour une partie du récepteur des gouts sucrés. On parle dans ce cas-ci d’un pseudogène, d’un gène « brisé » qui crée un problème dans le fonctionnement du récepteur. Les oiseaux sont eux aussi dépourvus de la capacité de percevoir le sucré, car ils ont carrément perdu le gène qui code pour une partie du récepteur du sucré. Celui-ci ne se retrouve plus dans leur génome. Ce qui est intéressant, c’est que certains groupes d’oiseaux ont regagné la capacité de percevoir le sucré. Nous allons voir comment cela est possible au niveau cellulaire et ce qui s’est passé au niveau évolutif. Finalement, outre les félins, la majorité des mammifères ont la capacité de détecter le sucré, mais de nouvelles approches de génomique nous permettent de croire que ce n’est pas le cas de toutes les espèces de ce groupe. Nous verrons l’exemple des baleines.
3.2 Un pseudogène pour un récepteur du gout chez les félins
Les félins sont des carnivores et plusieurs études comportementales ont démontré leur manque d’intérêt pour les aliments sucrés, tout en ayant des réponses similaires aux autres mammifères envers le salé, l’acide, l’amer et la présence d’acides aminés. Des biologistes se sont intéressés à cette absence de perception du sucré et ont proposé deux hypothèses. La première est qu’une mutation est apparue qui a créé un allèle de « non-gouteur », similaire aux individus qui ne perçoivent pas l’amertume du glucosinolate quand ils sont homozygotes pour l’allèle AVI. Si c’est le cas, leur prédiction était qu’on retrouverait une séquence du gène codant pour le récepteur du sucré, mais qu’il contiendrait une mutation qui affecte la séquence d’acides aminés de la protéine. La deuxième hypothèse proposée était que le gène Tas1r2 codant pour la protéine T1R2 qui forme un dimère avec la protéine T1R3 pour détecter le sucré est un pseudogène. La prédiction qui découle de cette deuxième hypothèse est que le gène serait tronqué et ne coderait plus pour une protéine complète. On peut donc assez facilement tester ces deux prédictions en déterminant la séquence de ces gènes chez les félins.
Les biologistes ont comparé la séquence des gènes qui codent pour les protéines T1R2 et T1R3 qui ensemble forment le récepteur pour le sucré, et ce pour différentes espèces : le chat, l’humain, le chien, la souris et le rat. En étudiant la séquence codante, elles ont découvert une délétion de 247 paires de bases chez le chat dans un exon de T1R2, alors que le gène T1R3 du chat était similaire à celui des autres mammifères étudiés (figure 17.17). Elles ont aussi trouvé cette délétion chez le tigre et le guépard. Cette délétion crée un changement de cadre de lecture et elles ont donc pu prédire que la traduction en acides aminés donnerait une protéine différente à partir de cette délétion. Le nouveau cadre de lecture entrainait aussi un codon stop avant la fin de la protéine chez le chat. Si une protéine est traduite à partir de cette séquence, elle serait donc formée d’acides aminés différents et serait tronquée comparativement à celle des autres mammifères étudiés. Les biologistes ont de plus trouvé 4 autres codons stop plus loin dans la séquence. Elles en sont venus à la conclusion que chez les félins, le gène T1R2 est un pseudogène. De plus, lorsqu’elles ont tenté de localiser l’expression de ce gène dans les papilles de chat à l’aide d’hybridation in situ, elles n’ont observé aucune expression, bien que leur méthode permette de détecter l’expression de l’autre moitié de l’hétérodimère, soit T1R3 [44].
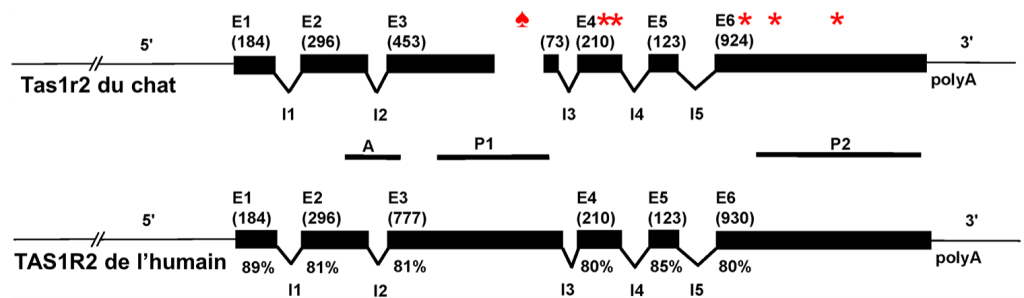
Les biologistes qui ont fait cette découverte dans le génome des chats ont suggéré que ceci représente une relaxation de la sélection naturelle sur ce gout lié à la diète exclusivement carnivore des félins. Comme la dérive génétique est toujours en action dans toutes les populations, peu importe leur taille, on peut émettre l’hypothèse que sans force de sélection pour la contrer, la dérive génétique par accumulation de mutation a résulté en ce phénotype particulier aux félins.
3.3 Perte d’un gène codant pour un récepteur du gout chez les oiseaux
L’étude du génome d’une espèce nous permet de faire d’importantes découvertes pour comprendre l’évolution des systèmes physiologiques, comme le montre l’étude sur les félins. Ainsi, l’étude du génome de différentes espèces d’oiseaux démontre que l’ancêtre de ce groupe a perdu le gène codant pour T1R2 dans son génome. En effet, contrairement aux félins qui ont un gène similaire à la protéine T1R2 des autres mammifères, mais une version non fonctionnelle, les oiseaux en général n’ont tout simplement pas ce gène dans leur génome. Une analyse du génome des alligators montre que ce gène y est présent, ce qui suggère que la perte du gène chez les oiseaux s’est produite après la séparation de ces deux groupes, tôt dans l’évolution des oiseaux [45, 46]. Ceci suggère que les oiseaux en général ne peuvent percevoir le gout sucré. Cependant, il existe plusieurs groupes d’oiseaux qui ont une diète composée de nectar ou de fruits, comme les colibris, les perroquets et de nombreux passériformes, spécialement dans le sous-ordre des oiseaux-chanteurs (les oscines). Ceci a amené des biologistes en écologie sensorielle à s’intéresser à l’évolution du gout du sucré chez ces oiseaux, à la suite de la perte du gène T1R2. Ils se sont demandé si ces groupes d’oiseaux ont un gène différent dans leur génome qui code pour un récepteur permettant de percevoir le sucré, ce que l’on peut prédire basé sur leur diète.
3.4. Mutation dans la séquence codante et évolution de la fonction d’un récepteur du gout chez les oiseaux
Les biologistes ont découvert que le gout sucré est détecté chez certains oiseaux par un récepteur qui existait déjà et qui a changé de fonction par des mutations qui sont apparues dans le génome puis a été sous sélection. L’hypothèse est que ces changements de fonction du récepteur apportaient un meilleur fitness et que le phénotype a donc été favorisé par la sélection naturelle. La mutation est dans la séquence codante du gène qui code pour un récepteur du gout. Ce qui est intéressant est que l’évolution de la perception du gout sucré est arrivée au moins deux fois chez les oiseaux : chez les colibris et les oiseaux-chanteurs. Est-ce que l’évolution a pris le même chemin deux fois?
Chez les oiseaux en général, les gènes T1R1 et T1R3 forment un dimère qui permet de détecter l’umami, comme chez d’autres vertébrés. Des biologistes ont démontré que chez les colibris, des mutations ont eu lieu il y a entre 22 et 42 millions d’années et ont été conservées. En effet, la séquence du gène codant pour la protéine T1R3 a divergé rapidement de celle des autres oiseaux pour lesquels les chercheurs et chercheuses avaient des séquences à comparer, notamment le martinet qui est une espèce proche des colibris (figure 17.18). Ils ont pu reconstruire la séquence ancestrale présente chez l’ancêtre commun des martinets et des colibris (figure 17.18, « AR1 ») ainsi que la séquence ancestrale des colibris après leur séparation des martinets (figure 17.18, « AR2 »). Cette divergence de séquence semble être sous sélection.
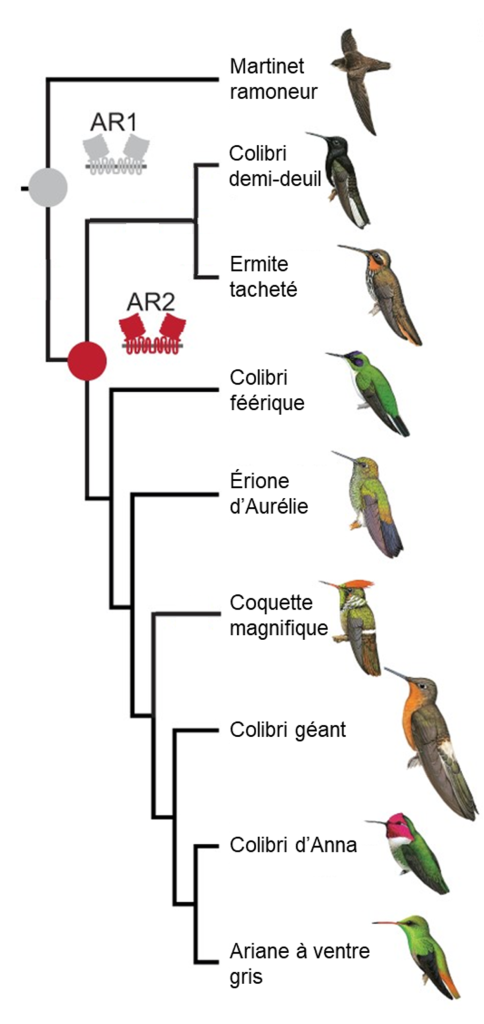
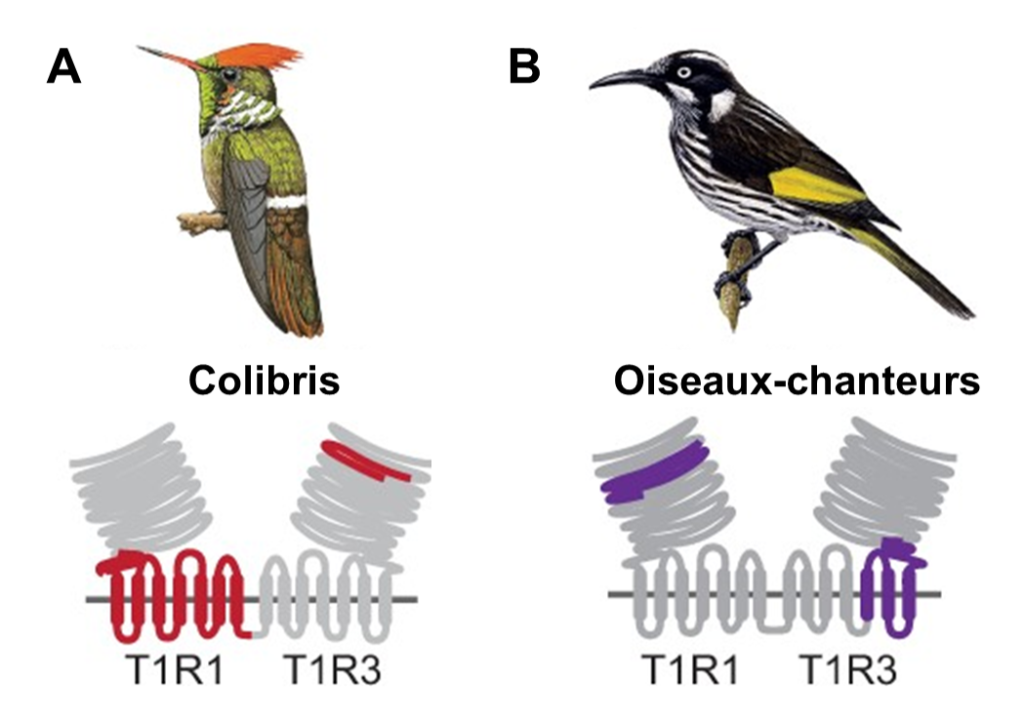
Évidemment, trouver une mutation nous donne une piste de recherche, mais ce n’est pas une preuve suffisante que celle-ci a effectivement un effet sur le phénotype ni que ce phénotype affecte la performance et augmente le fitness. Les biologistes ont donc fait une analyse fonctionnelle qui a permis de faire directement le lien entre les mutations et leur effet sur le fonctionnement du récepteur. En utilisant des lignées cellulaires qui permettent de mesurer la concentration de calcium à l’intérieur de la cellule (un indicateur que le récepteur est activé, comme dans l’étude de la perception de l’amer des singes du Nouveau Monde), ils ont démontré que ces récepteurs T1R1 et T1R3 du colibri répondent au gout sucré ensemble, mais pas lorsqu’ils sont exprimés séparément. Ils ont aussi découvert que ce sont des sections spécifiques de la protéine T1R3 qui ont évoluées et que ces changements se trouvent dans le site de liaison du récepteur (figure 17.19A). De plus, leur analyse fonctionnelle a révélé que le récepteur gustatif ancestral de tous les colibris (AR2 dans la figure 17.18) répond fortement aux sucres, mais pas le récepteur gustatif ancestral de l’ancêtre commun des colibris et des martinets (AR1 dans la figure 17.18). Ceci indique que l’acquisition de la réponse au gout sucré du colibri a eu lieu après la divergence avec leur ancêtre commun d’avec les martinets. Les mutations dans T1R3 ont donc changé sa sensibilité, ajoutant le sucre à la perception déjà présente d’acides aminés (figure 17.19A) [45, 47].
Cette même équipe de recherche s’est aussi intéressée aux oiseaux-chanteurs. Ils ont testé si les mêmes mutations étaient la cause de la détection du sucré chez ce groupe d’oiseaux que chez les colibris. Ils ont comparé le génome de plusieurs espèces d’oiseaux chanteurs connus pour percevoir le sucré avec celui d’oiseaux de groupes évolutivement apparentés qui ne détectent pas le sucré. Les biologistes ont découvert que les mutations qui affectent le site de liaison du récepteur ont eu lieu dans la protéine T1R1 chez les oiseaux-chanteurs, au lieu de T1R3, comme chez les colibris (figure 17.19B). Les mutations ont aussi affecté les résidus dans la région du site de liaison avec la molécule sucrée. Le récepteur est maintenant sensible au sucré en plus de l’umami, par une autre voie évolutive que chez les colibris [46, 47]. Il y a donc évolution convergente de la perception du gout sucré chez ces deux groupes d’oiseaux, mais ceci passe par des voies cellulaires différentes.
4. Écologie inversée : traduire les informations sur le génome en information sur l’écologie
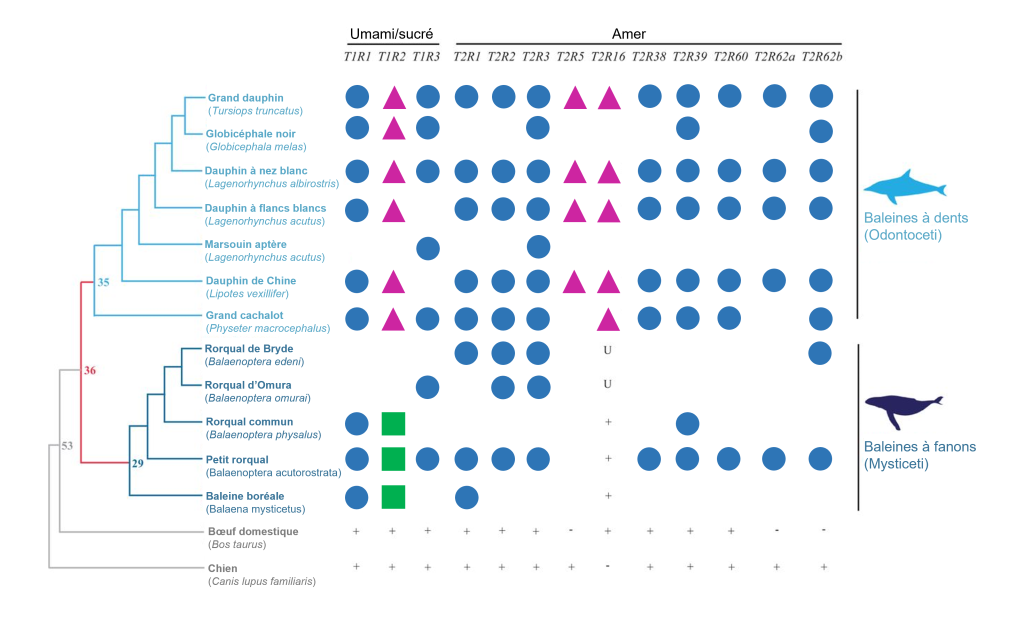
Nous avons vu que nous pouvons utiliser les séquences d’un génome pour prédire la perte de perception des gouts chez les vertébrés. Pour les espèces difficiles à étudier expérimentalement, l’étude du génome peut nous permettre d’en apprendre plus sur leur physiologie. Cette approche, qui s’appelle l’écologie inversée (de « reverse ecology » en anglais), consiste à commencer par l’étude du génome pour découvrir si des gènes sont présents et s’ils ont divergé pour mieux comprendre l’écologie d’une espèce et son interaction avec son environnement. Par exemple, les baleines sont difficiles à étudier afin de savoir quels gouts elles peuvent percevoir (même si de braves biologistes le font avec des études comportementales en milieu naturel!), mais on peut au moins scruter leur génome. Des biologistes se sont demandé si les baleines ont des gènes fonctionnels codant pour les différents récepteurs du gout. Elles ont étudié les génomes disponibles de 11 espèces de baleines (représentant les baleines à dents et les baleines à fanon), afin de déterminer la séquence des gènes de la famille T1R : T1R1 (umami), T1R2 (sucré), T1R3 (partagé pour former un hétérodimère) et 10 récepteurs de la famille T2R (amer) qui étaient connus pour être présents dans le génome d’un dauphin (figure 17.20). Elles ont aussi vérifié la séquence des gènes codant pour les récepteurs de l’acide et du salé chez 3 espèces. Leurs analyses ont permis de démontrer que tous les gènes de la famille T1R sont des pseudogènes (figure 17.20) [48].
Cas appliqué
Texte adapté de [49].
Les chauves-souris vampires sont les seuls mammifères qui se nourrissent exclusivement de sang. Cette spécialisation alimentaire exceptionnelle se reflète dans tous les aspects de leur biologie, notamment la morphologie, la physiologie et le comportement. Pour détecter ses proies, la chauve-souris vampire commune (Desmodus rotundus) présente un système olfactif bien développé, des capacités auditives avancées dans les basses fréquences et uniques parmi les mammifères, ainsi que la capacité de détecter le rayonnement infrarouge. Comparées aux autres chauves-souris, les chauves-souris vampires possèdent des capacités de locomotion terrestre exceptionnelles pour se faufiler et attaquer leurs proies. Leurs incisives supérieures tranchantes et sans émail aident à couper la peau de la proie, et les anticoagulants présents dans leur salive empêchent le sang de la proie de coaguler pendant l’alimentation.
En tant que seule source nutritionnelle, le sang représente un régime alimentaire qui apporte son lot de difficultés. Premièrement, le sang a une teneur élevée en liquide de 78 % et une valeur calorique relativement faible, ce qui oblige une chauve-souris vampire à ingérer jusqu’à 1,4 fois son poids corporel en sang au cours d’un seul repas. Pour permettre l’ingestion de grandes quantités de sang, leur estomac a connu un changement fonctionnel vers un organe extensible principalement utilisé pour le stockage et l’absorption des liquides. Deuxièmement, le sang a une teneur élevée en fer par rapport aux autres régimes alimentaires, qui provient principalement de l’hème dérivée de l’hémoglobine et du fer ferrique transporté par la transferrine. Troisièmement, la masse sèche de sang a une composition nutritionnelle très asymétrique, fournissant principalement des protéines (93 %) avec très peu de lipides et de glucides (1 % chacun). En raison de leur faible apport en glucides, les chauves-souris vampires présentent des niveaux d’insuline basale inférieurs à ceux des autres mammifères. Semblables aux patients humains diabétiques de type 2, les chauves-souris vampires présentent une réponse réduite de sécrétion d’insuline stimulée par le glucose, entrainant une hyperglycémie lors d’une surcharge expérimentale de glucose. Les réserves de glycogène et de lipides sont également réduites chez les chauves-souris vampires, ce qui contribue à leur vulnérabilité au jeûne et à leur mort précoce après 48 à 72 heures de jeûne. Les chauves-souris vampires partagent leur sang régurgité avec leurs compagnons de perchoir qui n’ont pas réussi à obtenir un repas nocturne ce qui peut compenser leur vulnérabilité au jeûne.
Pour découvrir les changements génomiques associés à cette adaptation alimentaire, des biologistes ont séquencé le génome de la chauve-souris vampire commune et comparé celui-ci au génome de 27 espèces de chauves-souris. Étant donné que la perte de gènes ancestraux peut constituer une force évolutive importante et que des études antérieures ont révélé de nombreuses associations entre les pertes de gènes et les différences phénotypiques, y compris les adaptations alimentaires, ils ont effectué un criblage à l’échelle du génome à la recherche de gènes spécifiquement perdus dans la lignée des chauves-souris vampires.
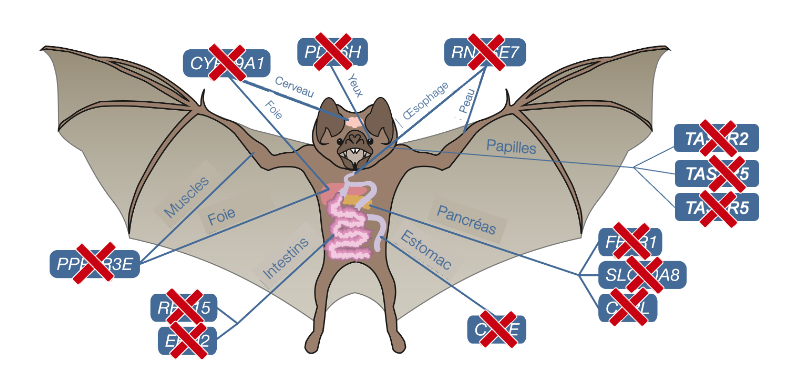
Ils ont découvert des pertes de gènes liées au mode de vie de cette espèce (figure 17.21). Ils ont observé la perte de gènes liés à une sécrétion réduite d’insuline (FFAR1 et SLC30A8 habituellement exprimés dans le pancréas), à des réserves limitées de glycogène (PPP1R3E habituellement exprimé dans le foie et les muscles) et à une physiologie gastrique unique (CTSE). D’autres pertes de gènes reflètent probablement la composition nutritionnelle biaisée (ERN2 et CTRL exprimés habituellement dans l’intestin) et la diversité distincte des agents pathogènes du sang (RNASE7). La perte de deux gènes leur suggère l’absence totale de vision basée sur les cônes chez ces chauves-souris strictement nocturnes (PDE6H et PDE6C qui sont habituellement exprimés dans la rétine). Ils ont aussi observé la perte des gènes qui codent pour des récepteurs du gout. En effet, le gène du récepteur du gout sucré TAS1R2 et les gènes du récepteur du gout amer TAS2R5 et TAS2R42 étaient absents du génome (habituellement exprimés dans les papilles gustatives).
Finalement, la perte de REP15, un gène impliqué dans la régulation de l’absorption cellulaire du fer, est probablement liée au régime sanguin obligatoire, riche en fer, des chauves-souris vampires. Malgré l’importance du fer pour divers processus cellulaires, une surcharge en fer peut avoir de graves effets néfastes. La chauve-souris vampire commune tolère des niveaux extrêmes de fer alimentaire sans présenter d’effets indésirables; la quantité relative de fer alimentaire a été estimée à 800 fois supérieure à celle des humains. Il faut tout de même un mécanisme pour éliminer le fer en surplus. Une étude sur la distribution du fer dans le tractus gastro-intestinal avait déjà révélé que D. rotundus utilise l’excrétion du fer comme mécanisme pour réduire les niveaux systémiques de fer. Plus précisément, on retrouve de grandes accumulations de fer dans les vacuoles contenant de la ferritine dans les cellules épithéliales gastro-intestinales de cette chauve-souris. Étant donné que l’épithélium intestinal a un temps de renouvèlement relativement rapide, l’excrétion des cellules gastro-intestinales contenant du fer dans la lumière intestinale élimine le fer du corps. En se basant sur toutes ces observations, les biologistes ont proposé que la perte de REP15 chez D. rotundus résulte en une augmentation de l’accumulation de fer dans les cellules du tractus gastro-intestinal et à son élimination. Les biologistes interprètent la perte de REP15 comme la perte de l’absorption de fer par les cellules, menant à l’élimination du surplus de fer par le tractus digestif.
Les gènes perdus au cours de l’évolution sont influencés par plusieurs facteurs. Un facteur important à l’origine de la perte de gènes est l’absence de sélection permettant de préserver la fonction de certains gènes à la suite de l’adaptation à un nouvel environnement ou à un mode de vie différent. Les gènes accumulent des mutations et deviennent des pseudogènes et sont éventuellement perdus, ou cette région du génome subit une délétion. Bien que l’aspect non indispensable de la fonction des gènes soit certainement la principale explication des pertes de gènes dans la lignée des chauves-souris vampires, les scientifiques ont proposé que la perte de gènes ancestraux puisse parfois être bénéfique dans l’évolution et contribuer aux adaptations, comme dans le cas de REP15.
Résumé
Au moins cinq types de gout sont perçus par les vertébrés grâce à des récepteurs associés à des cellules épithéliales connectées au système nerveux sensoriel qui mène ces informations au système nerveux central. Ces récepteurs perçoivent des molécules de deux façons : ils sont soit des récepteurs à canal ionique ou des récepteurs couplés à une protéine G. Les récepteurs de l’amer de la famille T2R ont évolué en de nombreuses copies à l’intérieur d’un génome donné par duplication suivi de néo-fonctionnalisation. Il y a des différences importantes dans le nombre de gènes codant pour un récepteur de l’amer entre les espèces. On peut étudier si la divergence de séquences entre les paralogues codants pour des récepteurs (le même récepteur entre deux espèces et différents récepteurs de la famille T2R) affecte la sensibilité à différentes molécules amères grâce à une expérience in vitro où on mesure le flux de calcium dans une cellule qui exprime un récepteur d’intérêt. On peut utiliser une analyse phylogénétique pour prédire la séquence ancestrale du récepteur avant sa divergence entre les espèces d’un même groupe. Chez les humains, on observe un cas de variation interindividuelle de sensibilité à une molécule amère, le glucosinolate, et la mutation dans la séquence codante du gène qui cause cette différence de sensibilité a été identifiée. Dans le cas du sucré et de l’umami, les récepteurs font partie d’une famille (T1R) dont les séquences sont très conservées évolutivement. Ces récepteurs sont formés d’un dimère de deux protéines distinctes. Lorsque la protéine T1R3 forme un dimère avec la protéine T1R2, la structure détecte le sucré. Lorsque T1R3 forme un dimère avec T1R1, le récepteur résultant détecte l’umami. Certains groupes de vertébrés ne perçoivent pas le sucré. Le gène codant pour la protéine formant le récepteur du sucré est non fonctionnel chez les félins à la suite d’une pseudogénisation. Dans le cas des oiseaux, l’absence de détection du sucré est liée à une perte d’un gène. Chez un groupe spécifique d’oiseau, les colibris, il y a eu « ré-évolution » de la perception du sucré par des mutations de novo qui ont changé la sensibilité à l’umami vers une sensibilité au sucré dans la protéine T1R3 qui est la protéine qui forme le dimère avec T1R1 et T1R2. Chez un autre groupe d’oiseau qui perçoit aussi le sucré, c’est plutôt une mutation dans le gène qui code pour la protéine T1R1 qui a ajouté la sensibilité aux molécules sucrées en plus des molécules umami. On peut utiliser les données de séquençage de génome pour étudier des traits physiologiques chez des espèces qui sont difficiles à étudier, comme pour prédire la sensibilité à différents gouts chez des mammifères marins.
Activité de révision
Exercices
Révision sur l’évolution du gout
Bibliographie
- Biga, L.M., S. Bronson, S. Dawson, A. Harwell, R. Hopkins, J. Kaufmann, M. LeMaster, P. Matern, K. Morrison-Graham, K. Oja, D. Quick, J. Runyeon, O. OERU, and OpenStax (2019). Taste, in Anatomy & Physiology [CC-BY-SA]. Oregon State University. https://open.oregonstate.education/aandp/chapter/15-1-taste/.
- Betts, J.G., P. Desaix, E. Johnson, J.E. Johnson, O. Korol, D. Kruse, B. Poe, J.A. Wise, M. Womble, and K.A. Young (2022). The Tongue, in Anatomy and Physiology 2e. OpenStax. [CC-BY]. https://openstax.org/books/anatomy-and-physiology-2e/pages/14-1-sensory-perception.
- Bradbury, J. (2004). Taste Perception: Cracking the Code PLOS Biology. 2(3): p. e64. https://doi.org/10.1371/journal.pbio.0020064.
- Betts, J.G., P. Desaix, E. Johnson, J.E. Johnson, O. Korol, D. Kruse, B. Poe, J.A. Wise, M. Womble, and K.A. Young (2022). The Brain Stem, in Anatomy and Physiology 2e. OpenStax. [CC-BY]. https://openstax.org/books/anatomy-and-physiology-2e/pages/13-2-the-central-nervous-system.
- Ahmad, R. and J.E. Dalziel (2020). G Protein-Coupled Receptors in Taste Physiology and Pharmacology [CC-BY]. Frontiers in Pharmacology. 11. https://www.frontiersin.org/articles/10.3389/fphar.2020.587664.
- Ramsey, I.S. and J.A. DeSimone (2018). Otopetrin-1: A sour-tasting proton channel Journal of General Physiology. 150(3): p. 379-382. https://doi.org/10.1085/jgp.201812003.
- Henley, C. (2021). Salt Taste Transduction, in Foundations of Neuroscience [CC-BY-NC-SA]. https://openbooks.lib.msu.edu/neuroscience/chapter/taste/.
- Henley, C. (2021). Sour Taste Transduction, in Foundations of Neuroscience [CC-BY-NC-SA]. https://openbooks.lib.msu.edu/neuroscience/chapter/taste/.
- Henley, C. (2021). Sweet Taste Transduction, in Foundations of Neuroscience [CC-BY-NC-SA]. https://openbooks.lib.msu.edu/neuroscience/chapter/taste/.
- Henley, C. (2021). Umami Taste Transduction, in Foundations of Neuroscience [CC-BY-NC-SA]. https://openbooks.lib.msu.edu/neuroscience/chapter/taste/.
- Henley, C. (2021). Bitter Taste Transduction, in Foundations of Neuroscience [CC-BY-NC-SA]. https://openbooks.lib.msu.edu/neuroscience/chapter/taste/.
- Shi, P. and J. Zhang (2005). Contrasting Modes of Evolution Between Vertebrate Sweet/Umami Receptor Genes and Bitter Receptor Genes Molecular Biology and Evolution. 23(2): p. 292-300. https://doi.org/10.1093/molbev/msj028.
- Freund, J.R., C.J. Mansfield, L.J. Doghramji, N.D. Adappa, J.N. Palmer, D.W. Kennedy, D.R. Reed, P. Jiang, and R.J. Lee (2018). Activation of airway epithelial bitter taste receptors by Pseudomonas aeruginosa quinolones modulates calcium, cyclic-AMP, and nitric oxide signaling Journal of Biological Chemistry. 293(25): p. 9824-9840. https://www.sciencedirect.com/science/article/pii/S002192582039966X.
- Köhler, F.E. (1897). Cinchona calisaya – Köhler’s Medizinal Pflanzen [Domaine public]. https://commons.wikimedia.org/wiki/File:Cinchona_calisaya_-_K%C3%B6hler%E2%80%93s_Medizinal-Pflanzen-179.jpg?uselang=fr.
- Schorzman, J. (2005). A small cup of coffee [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:A_small_cup_of_coffee.JPG?uselang=fr.
- Pimthida (2013). Tea 2 [CC-BY-NC-SA]. https://www.flickr.com/photos/pimthida/8502201742/in/photolist-dXj1nd-7kDf5F-pSLVBx-d6SFfo-3bbYM-dXdkaB-c8BVYf-dHYyGp-44L6S-qc54Kg-aE9fyW-MFd7QY-9adfNR-6hexMq-q84VjL-peaYWX-RAMHh8-8V5RDd-8WJLCa-ehbnS-7J9Mjv-KCoKL-5Ldjmf-w4Dntt-9fCi7s-dSBWij-wmz5UK-9Es8Ey-62Ym3W-w4yCjS-yaCa6y-82gPw-9kMNVN-c2UVkw-9yEcUg-vpfr3a-2pQBCP-6U3Get-nKoH4D-wmzxpP-aymrFq-4qKupH-cnPXFL-7Ugkt4-9cS9zi-j8BC2N-5hLz3z-J8uMdS-7Q6Sn2-5fp33F.
- formulatehealth (2020). Cocoa Powder and Chocolate on Marble Background [CC-BY]. https://commons.wikimedia.org/wiki/File:Cocoa_Powder_and_Chocolate_on_Marble_Background.jpg.
- Aubin-Horth, N. (2023). Enfant mangeant du yogourt [CC-BY-NC-SA].
- Hao, X., H. Jiao, D. Zou, Q. Li, X. Yuan, W. Liao, P. Jiang, and H. Zhao (2023). Evolution of bitter receptor genes and ontogenetic dietary shift in a frog [CC-BY-NC-ND]. Proceedings of the National Academy of Sciences. 120(8): p. e2218183120. https://www.pnas.org/doi/abs/10.1073/pnas.2218183120.
- Lei, W., A. Ravoninjohary, X. Li, R.F. Margolskee, D.R. Reed, G.K. Beauchamp, and P. Jiang (2015). Functional Analyses of Bitter Taste Receptors in Domestic Cats (Felis catus) PLOS ONE. 10(10): p. e0139670. https://doi.org/10.1371/journal.pone.0139670.
- Behrens, M., T. Lang, and S.I. Korsching (2023). A singular shark bitter taste receptor provides insights into the evolution of bitter taste perception Proceedings of the National Academy of Sciences. 120(48): p. e2310347120. https://www.pnas.org/doi/abs/10.1073/pnas.2310347120.
- Hallin, J. and C.R. Landry (2019). Regulation plays a multifaceted role in the retention of gene duplicates [CC-BY]. PLOS Biology. 17(11): p. e3000519. https://doi.org/10.1371/journal.pbio.3000519.
- Shaw, L., C. Mansfield, L. Colquitt, C. Lin, J. Ferreira, J. Emmetsberger, and D.R. Reed (2018). Personalized expression of bitter ‘taste’ receptors in human skin [CC-BY]. PLOS ONE. 13(10): p. e0205322. https://doi.org/10.1371/journal.pone.0205322.
- Li, D. and J. Zhang (2013). Diet Shapes the Evolution of the Vertebrate Bitter Taste Receptor Gene Repertoire Molecular Biology and Evolution. 31(2): p. 303-309. https://doi.org/10.1093/molbev/mst219.
- Behrens, M., Di Pizio, A., Redel, U., Meyerhof, W., & Korsching, S. I. (2021). At the root of T2R gene evolution: recognition profiles of coelacanth and zebrafish bitter receptors. [CC-BY]. Genome Biology and Evolution, 13(1), evaa264. https://doi.org/10.1093/gbe/evaa264
- dsasso (2005). A Night Monkey [CC-BY-SA]. https://en.wikipedia.org/wiki/File:Panamanian_night_monkey.jpg.
- Sanches, D. (2006). Sagui de Tufos Brancos [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:SAGUI-DE-TUFOS-BRANCOS.jpg.
- Tsutsui, K., M. Otoh, K. Sakurai, N. Suzuki-Hashido, T. Hayakawa, T. Misaka, Y. Ishimaru, F. Aureli, A.D. Melin, S. Kawamura, and H. Imai (2016). Variation in ligand responses of the bitter taste receptors TAS2R1 and TAS2R4 among New World monkeys [CC-BY]. BMC Evolutionary Biology. 16(1): p. 208. https://doi.org/10.1186/s12862-016-0783-0.
- h., j. (2005). broccoli baby [CC-BY-NC-SA]. https://www.flickr.com/photos/jamiehazlitt/37491629.
- Risso, D.S., M. Mezzavilla, L. Pagani, A. Robino, G. Morini, S. Tofanelli, M. Carrai, D. Campa, R. Barale, F. Caradonna, P. Gasparini, D. Luiselli, S. Wooding, and D. Drayna (2016). Global diversity in the TAS2R38 bitter taste receptor: revisiting a classic evolutionary PROPosal [CC-BY]. Scientific Reports. 6(1): p. 25506. https://doi.org/10.1038/srep25506.
- Sandell, M.A. and P.A. Breslin (2006). Variability in a taste-receptor gene determines whether we taste toxins in food Curr Biol. 16(18): p. R792-4.
- Monniaux, D. (2007). Endive [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Endive_p1160063.jpg.
- thebittenword (2008). Turnip [CC-BY]. https://commons.wikimedia.org/wiki/File:Turnip_2622027.jpg.
- Jengod (2009). Radishes [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Radish_3371103037_4ab07db0bf_o.jpg.
- jetsun (2010). Daikon, Nara Prefecture, Japan https://commons.wikimedia.org/wiki/File:Daikon,_Nara_Prefecture,_Japan.jpg.
- Evan-Amos (2011). Kale Bundle [Domaine public]. https://commons.wikimedia.org/wiki/File:Kale-Bundle.jpg.
- Hudson, T. (2012). Purple eggplant [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Purple_Eggplant.JPG.
- Buelldm (2012). Sprouted Alfalfa [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Sprouted_Alfalfa.jpg?uselang=fr.
- Coyau (2014). Brassica oleracea var. botrytis [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Chou-fleur_02.jpg.
- jackmac34 (2015). Carrots basket [Licence pixabay]. https://pixabay.com/photos/carrots-basket-vegetables-market-673184/.
- claralieu (2020). Bok choy [CC-BY]. https://commons.wikimedia.org/wiki/File:Bok_choy_-1.jpg.
- Madmad1234 (2020). Potato [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Potato_potato.jpg.
- Hodan, G. (2023). Spinach [Domaine public]. https://www.publicdomainpictures.net/en/view-image.php?image=163436&picture=spinach-leaves.
- Li, X., W. Li, H. Wang, J. Cao, K. Maehashi, L. Huang, A.A. Bachmanov, D.R. Reed, V. Legrand-Defretin, G.K. Beauchamp, and J.G. Brand (2005). Pseudogenization of a Sweet-Receptor Gene Accounts for Cats’ Indifference toward Sugar PLOS Genetics. 1(1): p. e3. https://doi.org/10.1371/journal.pgen.0010003.
- Baldwin, M.W., Y. Toda, T. Nakagita, M.J. O’Connell, K.C. Klasing, T. Misaka, S.V. Edwards, and S.D. Liberles (2014). Evolution of sweet taste perception in hummingbirds by transformation of the ancestral umami receptor Science. 345(6199): p. 929-33.
- Toda, Y., M.-C. Ko, Q. Liang, E.T. Miller, A. Rico-Guevara, T. Nakagita, A. Sakakibara, K. Uemura, T. Sackton, T. Hayakawa, S.Y.W. Sin, Y. Ishimaru, T. Misaka, P. Oteiza, J. Crall, S.V. Edwards, W. Buttemer, S. Matsumura, and M.W. Baldwin (2021). Early origin of sweet perception in the songbird radiation Science. 373(6551): p. 226-231. https://www.science.org/doi/abs/10.1126/science.abf6505.
- Cockburn, G., M.C. Ko, K.R. Sadanandan, E.T. Miller, T. Nakagita, A. Monte, S. Cho, E. Roura, Y. Toda, and M.W. Baldwin (2022). Synergism, Bifunctionality, and the Evolution of a Gradual Sensory Trade-off in Hummingbird Taste Receptors Mol Biol Evol. 39(2).
- Feng, P., J. Zheng, S.J. Rossiter, D. Wang, and H. Zhao (2014). Massive losses of taste receptor genes in toothed and baleen whales Genome Biol Evol. 6(6): p. 1254-65.
- Blumer, M., T. Brown, M.B. Freitas, A.L. Destro, J.A. Oliveira, A.E. Morales, T. Schell, C. Greve, M. Pippel, D. Jebb, N. Hecker, A.-W. Ahmed, B.M. Kirilenko, M. Foote, A. Janke, B.K. Lim, and M. Hiller (2022). Gene losses in the common vampire bat illuminate molecular adaptations to blood feeding Science Advances. 8(12): p. eabm6494. https://www.science.org/doi/abs/10.1126/sciadv.abm6494.

