2. Causes et conséquences de la variation phénotypique
Introduction au sujet
Dans ce chapitre, nous verrons d’abord l’intérêt d’étudier une discipline comme l’écophysiologie évolutive. Cette discipline se base sur des sous-disciplines clés de la biologie: la physiologie, l’écologie et l’évolution. Nous verrons ensuite comment on définit un phénotype, comment on retrouve ces phénotypes à différents niveaux d’organisation biologique et l’importance de comprendre les causes et les conséquences de la variation phénotypique. Pour ce faire, nous ferons un rappel de la définition de la variation génétique et de la variation environnementale et comment on peut étudier leurs effets sur la variation phénotypique.
Objectifs spécifiques
À la fin de ce chapitre, vous serez en mesure de :
- Comprendre que la combinaison de différentes disciplines nous permet de répondre à de nouvelles questions
- Définir le concept de phénotype
- Définir le concept de niveau d’organisation biologique
- Définir le concept de variation phénotypique
- Expliquer les causes de la variation phénotypique
- Analyser le résultat d’une expérience de jardin commun afin de différencier les effets de la variation génétique et de la variation environnementale
- Expliquer les conséquences de la variation phénotypique
Notions clés
- Combiner l’étude de la physiologie, l’écologie et l’évolution
- Le phénotype
- Les niveaux d’organisation biologique
- Quantifier la variation phénotypique
- Les causes de la variation phénotypique
- La variation génétique
- Mutation d’un seul nucléotide
- Les variants structuraux
- Locus et allèles
- Les traits discrets
- Les traits quantitatifs
- La variation environnementale
- Expérience de jardin commun
- Les conséquences de la variation phénotypique
1. Combiner l’étude de la physiologie, l’écologie et l’évolution
Les grandes questions du 21e siècle sont bien souvent en lien avec la biologie. Plusieurs changements environnementaux rapides induits par les humains nous causent des problèmes (figure 2.1). Que ce soient les enjeux de perturbation de l’environnement par les humains (fragmentation et perte d’habitat, pollution, monocultures), les effets des changements climatiques, la récolte excessive d’espèces sauvages ou la présence d’espèces invasives, on voit la trace des humains dans plusieurs problèmes mondiaux. La perte de biodiversité, l’émergence de nouveaux pathogènes, ainsi que l’évolution de la résistance aux traitements élaborés par les humains pour se débarrasser de ces pathogènes et de différentes pestes, sont aussi des problèmes criants qui nécessiteront l’apport des biologistes pour trouver des solutions.

Pour répondre à ces grandes questions du 21e siècle, il faut combiner des données de plusieurs disciplines. Si on pense spécifiquement à la biologie, même pour une question qui peut sembler être reliée à la sous-discipline de l’écologie, les biologistes savent maintenant que les connaissances en physiologie et en évolution doivent être prises en compte. Prenons l’exemple d’une espèce introduite par les humains dans un nouvel environnement, comme les cerfs de Virginie qui ont été introduits sur l’ile d’Anticosti au Québec à partir d’une population du continent (figure 2.2). Les biologistes s’intéressent à l’impact de cette population de cervidés sur l’écosystème insulaire et aussi à la divergence de ces cerfs en termes de taille, de réserves énergétiques, de traits d’histoire de vie et d’utilisation de l’habitat. Ils se demandent si ces cerfs ont subi de la sélection naturelle particulière en raison de l’environnement différent sur l’ile, menant à des changements de traits comme la morphologie, le comportement et la physiologie. En étudiant l’impact des individus sur la flore locale, ils ont constaté que la composition de la diète est différente sur l’ile comparativement à la diète des populations du continent au Québec. En effet, leur régime alimentaire est composé majoritairement de certains types de résineux, suite au déclin des feuillus résultant de leur herbivorie. Ces biologistes en sont venus à se demander si la physiologie digestive de ces animaux est modifiée en réponse aux conditions difficiles en hiver et, si c’est le cas, de quelle façon. Ils ont démontré que même la digestion est différente chez ces animaux, en plus des différences de taille et de réserves énergétiques. La capacité des cerfs à modifier leur physiologie digestive est potentiellement un facteur qui leur a permis de s’établir après leur introduction et à se maintenir une fois que leur présence a considérablement modifié les écosystèmes de l’ile.

Une autre grande question concerne les effets de la perturbation de l’habitat par les polluants. On peut étudier les traits qu’ils affectent, comme la reproduction et le comportement d’un individu, et comment ces changements se répercutent sur le fitness de l’individu et la croissance de la population. Cependant, pour vraiment comprendre l’impact de ces polluants, on doit aussi étudier comment ils causent ces dommages en se penchant sur les changements physiologiques et cellulaires qu’ils provoquent.
Si on s’intéresse aux effets des changements climatiques, on observe des variations de température et les effets de ces perturbations de l’environnement abiotique sur le comportement, la croissance ou même la survie des individus. En étudiant comment les systèmes physiologiques d’un organisme répondent à l’environnement qui change, on peut prédire ce qui arrivera aux individus d’une population dans différents scénarios de modification de l’environnement, comme une augmentation de la température. De plus, on peut se demander si les systèmes physiologiques vont pouvoir réagir adéquatement si l’environnement est modifié à un point tel que la population de cet organisme fait face à des conditions environnementales qu’elle n’a jamais rencontrées. Dans les deux cas, c’est la physiologie qui permet de comprendre le lien entre les changements de l’environnement et le succès des individus d’une population, une question centrale en conservation. Toutes ces questions nécessitent de combiner des approches pour y répondre et doivent être posées dans le cadre théorique de l’écophysiologie évolutive.
2. Le phénotype
En écophysiologie évolutive, nous étudions plusieurs types de traits chez les organismes qui nous intéressent. Un trait est une caractéristique d’un individu, tel que la couleur d’un pelage ou le taux métabolique. On peut avoir affaire à un trait discret ou à un trait quantitatif, comme nous verrons dans les sections suivantes. Un trait peut prendre différentes valeurs, qu’on appelle le phénotype. En effet, un phénotype est une caractéristique observable et mesurable d’un individu. Dans le cas de notre pelage, le phénotype serait la couleur observée de notre organisme, tel que blanc ou brun. Pour le métabolisme, le phénotype serait la quantité d’oxygène utilisée par minute.
Un phénotype peut être mesuré directement, comme la forme du feuillage d’une plante, ou le nombre de rejetons d’un oiseau (figure 2.3). On peut aussi observer et mesurer des phénotypes qui ne sont pas visibles à l’œil nu, tel que la forme d’une enzyme dans une cellule ou la quantité d’ARN messagers (figure 2.3). Certains biologistes vont parler du phénotype d’un individu en faisant référence à la valeur de plusieurs traits chez cet individu qui forment une « combinaison ».

Voici une liste non exhaustive des phénotypes que nous pouvons étudier chez un individu.
| Morphologie | Apparence | Comportement |
|---|---|---|
|
|
|
| Trait d’histoire de vie | Physiologie | Composantes cellulaires |
|
|
|
Exercices
Vérifiez votre compréhension du concept de phénotype avec ce quiz formatif.
Est-ce un phénotype?
3. Les niveaux d’organisation biologique
Les phénotypes que nous étudions peuvent être classés dans différents niveaux d’organisation biologique, allant de la molécule jusqu’à l’organisme entier. Un niveau d’organisation biologique représente une unité de complexité biologique : les molécules, les cellules, les tissus, les organes, les systèmes physiologiques et, finalement, l’organisme (figure 2.4). Le niveau organismique est sous-divisé en niveaux morphologique, comportemental et d’histoire de vie. Ces différents niveaux interagissent et s’influencent directement et indirectement les uns les autres et conduisent à l’organisme que nous étudions.
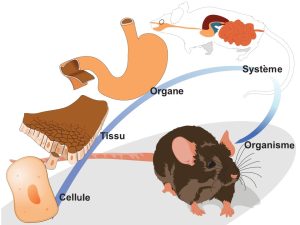
Ainsi, si nous avons pour objectif d’étudier un trait physiologique comme le métabolisme et comment il est modifié chez les organismes dont l’habitat est une rivière à courant rapide ou chez d’autres organismes qui volent en haute altitude, nous devons connecter des informations qui vont des molécules aux systèmes physiologiques (métabolisme, système musculaire, système nerveux, système respiratoire, système circulatoire), jusqu’à l’organisme entier.
Cette vidéo présente les niveaux d’organisation biologique.
Amoeba Sisters. (2017). Biological Levels in Biology: The World Tour [Vidéo]. Youtube. https://www.youtube.com/watch?v=EtWknf1gzKo&ab_channel=AmoebaSisters
Exercices
Vérifiez votre compréhension des niveaux d’organisation biologique avec ce quiz formatif.
Les niveaux d’organisation biologique
4. Quantifier la variation phénotypique
Lorsque nous observons le monde autour de nous, que ce soit avec nos yeux, un microscope ou une analyse moléculaire, nous voyons qu’il n’est pas uniforme. Nous remarquons bien sûr toute la diversité présente entre les espèces. Mais, même à l’intérieur d’une population, on observe des variations entre les individus. On parle alors de variation phénotypique et celle-ci peut être quantifiée. On peut par exemple classer des individus dans deux catégories si le trait est binaire, en les classant selon qu’ils présentent un phénotype ou un autre (« noir » ou « blanc », « présent » ou « absent », figure 2.5 panneaux a, e, f). Si le phénotype est une variable continue, on peut quantifier cette variation phénotypique en présentant un graphique de distribution de fréquence des différentes valeurs trouvées dans la population (figure 2.5 panneaux b, c, d). La variation phénotypique, ainsi que ses causes et ses effets, seront le sujet de toute l’unité 1.

5. Les causes de la variation phénotypique
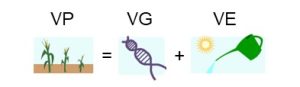
Lorsque nous observons et quantifions de la variation phénotypique, nous pouvons nous demander « qu’est-ce qui cause cette variation? ». Une explication pourrait être que les individus qui diffèrent dans la valeur d’un trait portent des mutations qui affectent le phénotype. Cette variation du phénotype pourrait aussi être le résultat de l’effet de l’environnement sur l’organisme. En fait, il est fort probable que ce soit un mélange des deux : la variation phénotypique résulte très souvent de la variation génétique et de la variation environnementale (Figure 2.6).
6. La variation génétique
On peut définir la variation génétique comme toutes les différences dans la séquence d’ADN d’un génome entre des organismes. On peut comparer des individus d’une même population, des individus de différentes populations de la même espèce, ou des individus classés comme différentes espèces. La variation génétique prend plusieurs formes. Elle peut se manifester comme un polymorphisme à un seul nucléotide ou être présente sur une plus grande région d’ADN, ce qu’on appelle des variants structuraux. La variation génétique est très importante à considérer lorsqu’on veut étudier l’évolution des adaptations physiologiques. C’est un concept central lorsqu’on étudie les forces évolutives, la réponse à la sélection et les adaptations.
6.1 Mutation d’un seul nucléotide
Une mutation peut se trouver dans la séquence codante d’un gène ou bien dans la séquence régulatrice d’un gène. Bien sûr, elle peut aussi se trouver à un endroit du génome qui n’est ni une séquence codante ni une séquence régulatrice.
Une séquence codante est la séquence du gène qui code pour la protéine. Il existe plusieurs formes de mutations qui peuvent se produire dans une séquence codante. On les divise en deux catégories selon leurs effets : les substitutions synonymes et non synonymes, selon la façon dont elles affectent un codon.
Les substitutions synonymes sont des mutations silencieuses, car la protéine reste la même. Le changement de nucléotide n’entraine aucun changement d’acide aminé, car il résulte en un nouveau codon qui une fois traduit donne le même acide aminé. Par exemple, GCU, GCC, GCA, GCG sont des codons différents, mais codent tous pour une sérine.
Les substitutions non synonymes peuvent être de deux types : les mutations faux-sens et les mutations non-sens. Dans le cas d’une mutation faux-sens, le changement de codon entraine la traduction d’un acide aminé différent. Quand on pense à « mutation », on pense souvent à ce type spécifique de mutation avec un effet, c’est-à-dire une mutation qui change la chaine d’acides aminés qui forment la protéine. Il est important de noter que ce nouvel acide aminé aboutira peut-être en une modification de fonction de la protéine, mais pas nécessairement. Certaines substitutions non synonymes sont neutres et n’affectent pas le fitness, et certaines mutations peuvent être délétères ou avantageuses.
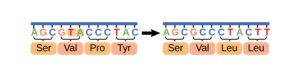
Cette vidéo présente les mutations faux-sens. Une région d’ADN est transcrite en ARN messager puis traduite en acides aminés. Une substitution à un nucléotide (A devient C) change le codon de UAC à UCC et celui-ci code maintenant pour un nouvel acide aminé (une sérine au lieu d’une tyrosine).
National Human Genome Research Institute. (2022). Missense mutation 3-D [Vidéo]. Youtube. https://www.youtube.com/watch?v=LoxOmFoQOQk&ab_channel=NationalHumanGenomeResearchInstitute
Dans le cas d’une mutation non-sens, le changement de nucléotide résulte en un nouveau codon qui est un codon-stop, ce qui « arrête » la synthèse de la protéine avant qu’elle ne soit terminée.
Cette vidéo présente les mutations non-sens. Une région d’ADN est transcrite en ARN messager puis traduite en acides aminés. Une substitution à un nucléotide (A devient C) change le codon de UAC à UAG et celui-ci code maintenant pour un codon-stop.
National Human Genome Research Institute. (2022). Nonsense mutation 3-D [Vidéo muette]. Youtube. https://www.youtube.com/watch?v=Qe05MxqNlJk&ab_channel=NationalHumanGenomeResearchInstitute
Exercices
Vérifiez votre compréhension de ces 3 types de mutations avec ce quiz formatif.
Mutation d’un nucléotide : types de substitution
Une séquence régulatrice est définie comme une séquence qui affecte l’expression d’un gène, c’est-à-dire qui a un effet sur la quantité de la protéine qui sera synthétisée, ainsi que sur le lieu et le moment de son expression. S’il y a une mutation dans cette séquence régulatrice, ça peut mener à un changement du niveau d’expression du gène. Cependant, la protéine elle-même ne sera pas différente.
6.2 Les variants structuraux
Les variants structuraux sont une autre grande classe de variation génétique. On classe divers types de réarrangements de l’ADN dans cette catégorie : les délétions et les insertions de nucléotide, les duplications d’une section d’ADN, les inversions et les translocations [21]. Les insertions et les délétions peuvent changer le cadre de lecture (sauf si l’insertion est un multiple de 3!). Ces deux types de variants structuraux peuvent avoir des effets très importants sur la structure de la protéine, car les acides aminés traduits seront différents lorsque le cadre de lecture sera modifié (figure 2.7).
Cette vidéo présente les changements de cadre de lecture. Une région d’ADN est transcrite en ARN messager puis traduite en acides aminés. Une insertion ajoute un nucléotide (C), ce qui change le cadre de lecture. Le codon passe de CAA qui code pour une glycine à CAC qui code maintenant pour une histidine.
National Human Genome Research Institute. (2022). Frameshift mutation 3-D [Vidéo muette]. Youtube. https://www.youtube.com/watch?v=yv_vnMKdJzY&ab_channel=NationalHumanGenomeResearchInstitute
Les autres variants structuraux sont des altérations génomiques de plus grande taille, habituellement définies comme ayant au moins 50 paires de bases. Les duplications de séquence peuvent être sous la forme d’une ou plusieurs copies d’une certaine portion d’ADN, qui peuvent contenir ou non la région d’un gène. Les variations du nombre de copies (souvent appelé « CNV » pour copy number variation) sont un sous-type particulier de variants structuraux. Elles résultent de la duplication d’une région (image 2.8).
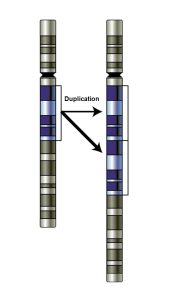
Cette vidéo présente la duplication. Une région d’ADN (AATT) est dupliquée plusieurs fois et forme maintenant une chaine de nucléotides AATTAATTAATTAATT. Une duplication peut aussi se produire dans une large région chromosomique.
National Human Genome Research Institute. (2022). Duplication 3-D [Vidéo muette]. Youtube. https://www.youtube.com/watch?v=dHdA3m4zeuM&ab_channel=NationalHumanGenomeResearchInstitute
Finalement, les inversions et translocations, comme leur nom l’indique, sont des séquences d’ADN qui sont inversées ou déplacées dans le génome par rapport à la séquence originale.
Cette vidéo présente les inversions. Une représentation de chromosome schématique montre qu’une région d’ADN peut s’inverser.
National Human Genome Research Institute. (2022). Inversion 3-D [Vidéo muette]. Youtube. https://www.youtube.com/watch?v=nPtTEiNrJSM&ab_channel=NationalHumanGenomeResearchInstitute
6.3. Locus et allèle
Un locus désigne une région de l’ADN qui peut constituer une position spécifique à un seul nucléotide, contenir un gène ou encore une plus longue région. Un allèle est la séquence de nucléotides à ce locus. Par exemple, une mutation d’un nucléotide d’un G vers un C à une position du génome mènera à deux allèles possibles à ce locus. S’il y a une autre mutation à la même position (disons un A), il y aura 3 allèles dans la population à ce locus. Nous discuterons principalement des eucaryotes, qui sont la plupart du temps diploïdes. Les organismes diploïdes ont deux chromosomes pour les autosomes (chromosomes non sexuels). L’allèle peut être le même pour les deux copies à ce locus et on parle alors de génotype homozygote. L’organisme peut plutôt avoir des allèles différents pour chaque copie à ce locus, ce qui représente un génotype hétérozygote. Dans une population, on peut retrouver plusieurs allèles à un locus : par exemple, s’il existe 6 allèles différents, un individu possèdera une certaine paire de ces 6 allèles. Le ou les allèles qui composent le génotype d’un individu à un locus peuvent affecter son phénotype (ou non).
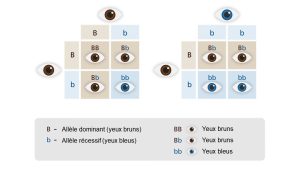
Pour préciser que la présence d’un allèle entraine un phénotype particulier, peu importe l’autre allèle qui compose le génotype diploïde, on parle d’un allèle dominant. L’allèle qui affecte le phénotype seulement s’il est présent en deux copies (un génotype homozygote composé uniquement de cet allèle) est dit récessif. Par exemple, si on imagine un locus qui affecte la couleur des yeux, avoir un génotype qui inclut l’allèle dominant « B » en une ou deux copies (Bb ou BB) donnera des yeux bruns et on ne voit des yeux bleus que lorsque l’allèle récessif « b » est présent en deux copies, résultant en un génotype homozygote bb (figure 2.9). Dans l’exemple de gauche sur la figure, deux individus aux yeux bruns peuvent être tous les deux hétérozygotes (Bb) et avoir une certaine probabilité d’avoir un rejeton qui sera homozygote pour l’allèle récessif bb, résultant en des yeux bleus.
Exercices
Vérifiez votre compréhension du concept de dominance avec ce quiz.
Dominance et récessivité. Adapté de [25].
6.4. Les traits discrets
Pour un trait qui est affecté par le produit d’un seul gène, on va observer des valeurs discrètes qui vont varier selon l’allèle présent, par exemple, présence ou absence, ou des formes différentes sans intermédiaire. On peut penser à des maladies humaines comme l’anémie falciforme ou la fibrose kystique. Dans le cas de l’anémie falciforme, une personne portera un allèle et aura un globule rouge de type normal et une autre personne aura un autre allèle et aura un globule rouge d’un autre type (figure 2.10).
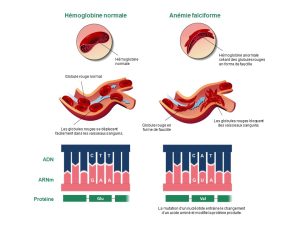
Dans ce cas précis, la maladie de l’anémie falciforme est apparente chez les homozygotes, donc qui ont deux allèles avec une mutation dans leur génotype. Pour des traits morphologiques comme la coloration du pelage des souris ou la présence ou l’absence d’épines chez les épinoches, on connait le gène dont le produit affecte le phénotype et on connait les variations génétiques associées à chaque valeur du trait.
Il existe de nombreux traits discrets pour lesquels nous n’avons aucune idée des bases génétiques. Malgré cela, on peut étudier ces traits et voir comment leur fréquence change dans la population, leur lien avec le fitness dans un environnement donné, etc. Les gens qui ont déterminé que les souris foncées avaient moins de chance de survie sur le sable pâle que des souris pâles, en raison de la prédation, n’avaient au départ aucune idée de l’influence du gène MC1R dans la coloration des souris, mais ils s’intéressaient tout de même à l’évolution de ce trait.
6.5. Les traits quantitatifs
Pour la plupart des traits, la valeur de ce trait est contrôlée par la variation du génotype pour plusieurs gènes. Par exemple, dans le cas de la taille d’un individu, on sait que plusieurs gènes contribuent à la différence de valeur observée entre les individus. Le génotype d’un individu à chacun de ces locus a une influence faible, mais réelle et, au total, on obtient la valeur de ce trait (figure 2.11). On parle alors d’un trait polygénique. La très grande majorité des traits physiologiques qui intéressent les biologistes travaillant en écophysiologie évolutive sont des traits polygéniques. Pour la plupart de ces traits, on ne connait pas l’identité des nombreux gènes qui sont impliqués dans les variations interindividuelles. Cependant, bien que ceux-ci soient plus rares, nous verrons plusieurs exemples où on connait un gène qui a un large effet, probablement parce que les traits qui sont polygéniques mais affectés en grande proportion par le génotype à un gène donné sont plus « faciles » à étudier et à découvrir!
Les traits polygéniques sont souvent appelés des traits quantitatifs. Nous reviendrons sur les traits quantitatifs lorsque nous discuterons de la réponse à la sélection. La génétique quantitative existe depuis longtemps et a été développée dans le cadre de l’amélioration par croisement d’organismes domestiqués, avant même qu’on ne connaisse les détails moléculaires qui sous-tendent l’hérédité. En effet, il est possible de faire des prédictions sur l’évolution des traits polygéniques, par exemple la quantité de lait que feront les descendants d’une vache ou la résistance à une maladie des descendants d’une plante, sans savoir quels gènes sont impliqués dans le trait qui nous intéresse!
7. La variation environnementale
Lorsqu’on observe des différences interindividuelles de phénotype, par exemple entre des individus d’une même population, une autre explication pour cette variation phénotypique pourrait être que ces individus ont fait face à des environnements différents durant leur développement. Si on y pense, nous pouvons tous imaginer un exemple de différences entre des individus d’une même population qui sont dues à l’environnement où se trouvaient ces organismes. Par exemple, des plantes se développent de façon très différente selon les conditions d’ensoleillement et de température, même si les graines proviennent toutes de la même plante (figure 2.12). On sait aussi que de nombreux changements ont lieu chez plusieurs animaux qui vivent en milieu tempéré selon la saison, comme les oiseaux qui entreprennent des migrations, les mammifères qui hibernent, ou encore les grenouilles qui vivent en milieu forestier l’hiver. Ces changements saisonniers de phénotypes sont entièrement dus à l’environnement et peuvent être majeurs. L’effet des différences dans l’environnement qui causent des différences de phénotypes est un aspect très important en physiologie et nous y reviendrons lorsque nous discuterons de l’acclimatation et de la plasticité phénotypique.

8. Expérience de jardin commun
Il existe une approche pour déterminer si la variation dans un trait est le résultat de la variation génétique ou la variation environnementale, ou les deux : c’est l’expérience de jardin commun. Cette expérience consiste à mettre les individus dans un environnement commun. Comme les premières expériences de ce type ont été faites sur des plantes, on les a appelées des expériences de jardin commun, mais on utilise aussi expérience en environnement commun.
On peut étudier une population où il y a des différences phénotypiques entre les individus. On reproduit les individus qui ont, par exemple, une taille élevée ensemble et ceux qui ont une taille faible ensemble. On élève tous leurs descendants dans un même milieu contrôlé, comme un laboratoire ou une serre. On peut ainsi les exposer à des conditions environnementales contrôlées et uniformes durant la période critique où le développement peut être affecté par l’environnement et quantifier le phénotype des différents rejetons (figure 2.13). Si on mesure des différences de phénotypes chez les rejetons des deux groupes, même sans différences de l’environnement, cette variation phénotypique peut être attribuée à des différences génétiques (bien que la cause même au niveau génétique reste inconnue). Si les rejetons provenant de parents qui avaient des différences de phénotypes ne montrent pas de différences significatives lorsqu’on maintient l’environnement contrôlé, on peut attribuer les différences observées en milieu naturel à de la variation environnementale. Bien sûr, une autre interprétation est que l’environnement choisi en milieu contrôlé n’affecte pas le développement, car il ne représente pas ce qui est vécu en nature. Il faut donc bien faire son design expérimental pour reproduire l’environnement original (ou les environnements originaux si les groupes proviennent d’environnements différents). Il faut donc parfois exposer les groupes à plus d’un environnement, en séparant les rejetons également entre l’environnement A et l’environnement B et ensuite quantifier leur phénotype.
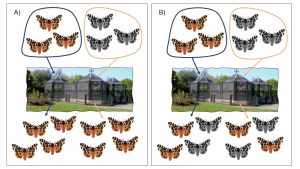
On pourrait aussi comparer des individus de plusieurs populations qui montrent des différences dans un phénotype, comme leur métabolisme à une certaine température. On ferait la même expérience en environnement commun, mais on comparerait les rejetons des différentes populations pour déterminer si les différences se maintiennent quand on enlève l’influence de l’environnement, ou lorsqu’on les fait se développer dans l’environnement auquel fait face l’autre population. Vous vous doutez bien que concrètement, on se rend le plus souvent compte que la variation phénotypique est le produit à la fois de la variation génétique et de la variation environnementale. Nous verrons comment quantifier la proportion de la variation phénotypique qui est attribuable à chaque facteur lorsque nous parlerons de l’héritabilité.
9. Les conséquences de la variation phénotypique
La variation phénotypique nous intéresse particulièrement parce qu’elle peut être associée à des différences de performance et de fitness. Prenons l’exemple de l’expérience classique des pinsons de Darwin : si on réunit dans une volière différentes espèces de pinsons de Darwin qui ont différentes formes et tailles de bec et qu’on les nourrit avec une sorte spécifique de nourriture (disons qu’ils ont tous une noisette comme source de nourriture), on pourra étudier leur capacité à se nourrir de ce type d’aliment. On peut ensuite répéter l’expérience avec différents aliments : fruits à coques, graines délicates, insectes. Il y aura une conséquence à la variation phénotypique: certains individus auront une meilleure performance d’alimentation que d’autres pour une certaine source de nourriture, mais pas pour une autre, selon leur morphologie qui affecte la force et la dextérité de leur bec, et ceci affectera leur survie si on continue l’expérience (figure 2.14). Dans ce cas-ci, on connait d’avance le résultat de l’expérience, mais dans bien des cas, le lien (ou l’absence de lien) entre les différences interindividuelles de phénotypes et les différences de fitness n’a pas été quantifié et on doit donc le vérifier.

Cas appliqué
Quantifier la variation phénotypique
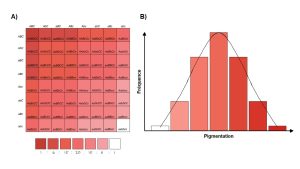
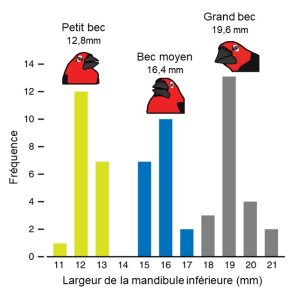
Des biologistes étudient la morphologie du bec chez le Pyréneste ponceau (Pyrenestes ostrinus) (figure 2.15). Le nom commun anglais de cette espèce est « le casse noix à ventre noir » et son bec a en effet une forme souvent associée à une capacité élevée de créer une forte pression.

L’étude de la morphologie de ces oiseaux nous donne un excellent exemple de distribution de fréquence de la valeur d’un phénotype (figure 2.16).
Mutation d’un seul nucléotide
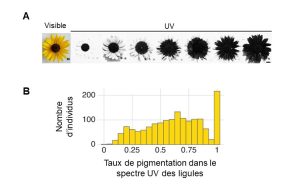
Des biologistes étudient le patron de pigmentation florale visible dans le spectre des UV chez une espèce de tournesol, Helianthus annuus. Ils observent de la variation interindividuelle dans le patron de pigmentation visible dans le spectre des UV (figure 2.17, panneau A). Chez le tournesol, les fleurs véritables, que nous voyons comme le cœur foncé de la plante, sont entourées de « pétales » formées en fait de ligules, qui sont jaunes à nos yeux. Les biologistes mesurent le degré de pigmentation visible dans le spectre des UV pour ces ligules, ce qui leur permet de créer une distribution de fréquence des différents phénotypes de pigmentation, allant d’aucune pigmentation des ligules à 100 % des ligules pigmentées (figure 17, panneau B).
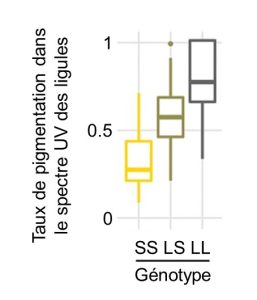
Les biologistes ont découvert un lien entre le génotype à un nucléotide en amont du gène HaMYB111 et la proportion de ligules pigmentés (figure 2.18). Des études antérieures chez d’autres espèces de plantes ont permis de découvrir que ce gène code pour un facteur de transcription qui contrôle l’expression d’autres gènes qui sont impliqués dans la production de molécules anti-oxydantes. Ces molécules ont aussi la propriété d’absorber les UV. Les biologistes ont déterminé qu’une mutation à un seul nucléotide à cette position dans le génome résulte en une différence de phénotype chez la plante. Les individus homozygotes pour l’allèle « S » ont une faible proportion de ligules pigmentées alors que les homozygotes pour l’allèle « L » ont de la pigmentation sur la plupart de leurs ligules (figure 2.18). Les hétérozygotes « SL » ont un phénotype intermédiaire. Grâce à des expériences de jardin commun, les biologistes ont démontré que ces différences de pigmentation sont maintenues que la plante soit élevée en milieu naturel ou en milieu contrôlé, suggérant qu’elles sont dues à de la variation génétique et excluant une influence de l’environnement.
Comment cette mutation a-t-elle un effet? Comme cette mutation est dans la séquence régulatrice du gène et non dans la séquence codante, la forme même de la protéine pour laquelle ce gène code est la même peu importe que la plante ait l’allèle S ou L, c’est plutôt la quantité de la protéine qui est exprimée qui diffère et affecte la pigmentation. Il y a ainsi des différences dans le niveau d’expression du facteur de transcription selon l’allèle, ce qui entraine une cascade d’effets sur les gènes dont les produits sont impliqués dans la synthèse des molécules anti-oxydantes, ce qui résulte en la pigmentation foncée dans le spectre de l’UV des tournesols.
Les variants structuraux
Des scientifiques étudient l’adaptation au stress de salinité chez le peuplier de l’Euphrate, Populus euphratica (figure 2.19). Pour ce faire, ils comparent le génome de ce peuplier qui vit en milieu salin avec celui d’une autre espèce de peuplier qui ne vit pas en milieu salin, le peuplier baumier de l’ouest (P. trichocarpa). Ils cherchent des différences génétiques entre les deux espèces qui pourraient être la cause de l’adaptation à la salinité du peuplier de l’Euphrate. Ils cherchent à quantifier la présence de variants structuraux, en testant si certains gènes sont en plus grand nombre de copie chez une des espèces de peuplier que chez l’ancêtre de cette espèce.

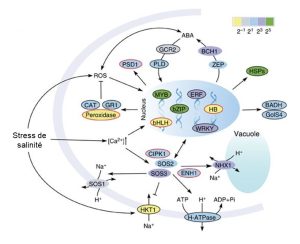
Les deux espèces de peuplier ont des gènes qui sont dupliqués comparativement à l’ancêtre commun. Cependant, le peuplier de l’Euphrate qui vit dans un milieu salin a un plus grand nombre de gènes qui ont un grand nombre de copies comparativement au peuplier baumier de l’ouest. Ces gènes sont associés à des fonctions cruciales pour la survie en milieu salin, tel que le transport des ions qui permet de réguler l’apport en sel (HKT1), ainsi que des gènes impliqués dans la création d’énergie pour soutenir les pompes qui contrôlent la quantité de sel dans les fluides de l’arbre (H-ATPases) (figure 2.20).
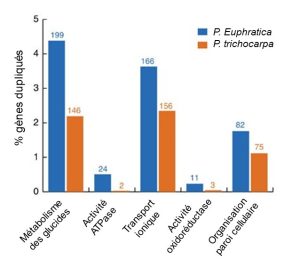
Ces gènes qui sont représentés en de nombreuses copies et qui sont impliqués dans la réponse au stress de salinité sont très exprimés lorsqu’on expose expérimentalement la plante à un stress de salinité (figure 2.21). Ils sont impliqués dans divers processus et se retrouvent dans le noyau, le cytosol et la membrane cellulaire.
Expérience de jardin commun
Des biologistes ont utilisé une expérience de jardin commun avec des sous-espèces d’oiseaux du genre Junco (figure 2.22) afin d’étudier les causes génétiques et environnementales de la variation de la capacité d’acclimatation au froid basée sur la capacité thermogénique, c.a.d. la capacité de produire de la chaleur.

Les biologistes ont étudié cinq populations de Junco qui font face à des régimes thermiques différents. Certaines se retrouvent dans des environnements stables et ne font pas face à de grandes variations en milieu naturel, alors que d’autres font face à de plus grande variation annuelle de température. Leur hypothèse est que le degré de variation annuelle affecte leur capacité d’acclimatation. Leur prédiction est que les populations vivant en milieu variable auront une plus grande capacité à s’acclimater et à produire de la chaleur face au froid.
Ils ont exposé les individus en laboratoire à différentes températures et ont mesuré le changement de consommation d’oxygène par minute, une estimation du métabolisme. Ils ont fait ces mesures avant et après une acclimatation de 3 semaines à deux températures (chaude (23C) et froide (3C)) (figure 2.23). Ils ont constaté que certaines populations ne changent pas leur consommation d’oxygène après 3 semaines d’acclimatation et ils les ont qualifiées de peu flexibles. D’autres populations montrent de grands changements, c.a.d. qu’elles ont une grande capacité de s’acclimater au froid et de consommer plus d’oxygène pour produire de la chaleur. Ces populations qui ont une plus grande capacité à s’acclimater et à produire de la chaleur face au froid sont celles qui vivent en milieu variable, tel que prédit. Comme ces différences inter-populations ont été mesurées en environnement commun, les biologistes ont suggéré que ces différences de capacité d’acclimatation résultent de la variation génétique.

Résumé
L’écophysiologie évolutive vise à étudier les causes et les conséquences de la variation phénotypique des systèmes physiologiques, à différents niveaux d’organisation. L’étude de ces causes vise à distinguer l’apport respectif de la variation génétique et de la variation environnementale à la biodiversité qui intéresse les biologistes, tout en permettant de répondre à de grandes questions d’actualité.
Activité de révision
Exercices
Révision sur la variation phénotypique
Bibliographie
- GRID-Arendal (2014). Cleared land in the Amazon Jungle, Brazil [CC-BY-NC-SA]. https://www.flickr.com/photos/gridarendal/31716767830/in/photostream/
- U.S. Department of Agriculture (2013). The Rim Fire in the Stanislaus National Forest near in California began on Aug. 17, 2013-0004 [CC-BY]. https://www.flickr.com/photos/41284017@N08/9599182665
- NIAID (2014). Ebola Virus From Mali Blood Sample [CC-BY]. https://www.flickr.com/photos/54591706@N02/15869044004
- Bárbol (2010). Tomorrow they will be gone… [CC-BY-NC-SA]. https://www.flickr.com/photos/10770266@N04/5061806365/in/photolist-8Hi5Fx-2jrkGe9-7Gzzbn-owi6xf-x3Jjn7-wKphkv-xiazRp-x1e8mv-xhTyBQ-xetrZC-tEwGH4-dTjj88-ouwMSt-xe4Fih-owmxuT-xLenLy-RfkmA1-sGSxYq-xe4hXL-wQwJhX-tkVvEg-Q57n72-y1wHYb-roRzCM-21Q4uM3-r7hxcu-xdUfzo-yezBpH-oupR9L-9LU73h-x1ripq-xZLLey-tnWAAG-ouu1Cj-xXNYhP-tnYYgx-y2sNou-xV8CoC-tEtBUj-wmi71M-ynsBEQ-wPhVfH-xhG6FP-ry5wzE-tnhZz4-tyN9rW-xtYVaB-xe9yvP-xfE7Nb-x3SPmt
- USFWS Mountain-Prairie (2022). Zebra Mussels (Dreissena polymorpha) [domaine public]. https://www.flickr.com/photos/usfwsmtnprairie/52115981907/in/photostream/
- U.S. National Oceanic and Atmospheric Administration (1969). Shrimp by-catch (Location: East Coast of w:Florida) [domaine public]. https://commons.wikimedia.org/wiki/File:Shrimp_bycatch.jpg
- Boucley, M. (2014). Les cerfs de l’île d’Anticosti au coucher du soleil [CC-BY-NC-SA]. https://monde.ccdmd.qc.ca/ressource/?id=107583&demande=desc
- Enzymlogic (2013). CDK8/cyclinC active conformation [CC-BY-SA]. https://www.flickr.com/photos/101755654@N08/11241208315
- Bergolli (2013). Bergolli_Konnevesi-poikue [CC-BY-NC]. https://www.flickr.com/photos/bergolli/9146612004/in/photolist-ovD4qg-of1Acz-8mZd5v-w7NB6h-7n62kG-wZ5yGZ-eWfM5Y-x4MNK2-cuSM7b-gcr8Ur-wMij8N-rpgagt-r6g6mt-rnhvLw-wMiiCu-owauXs-xFTwQD-qsybE9-x4Kzcp-wMbuuU-28HsRCJ-tkcCvi-D3HqPq-5fNwMo-351mDG-4mRJPJ-8EMP6f-6xWY3Q-2iVrpwc-6xWXS5-7y7bwK-wMibdQ-m1BUK4-w5SXy5-m1BSdF-m1BSHZ-m1BTi6-m1CvPF-m1Dmhy-oeW1c8-tkE2eQ-ovWSzV-an4EvS-sDGQaS-x5nsW2-r64NMP-x2vfKq-2ndnRXB-r6en4K-wMgiRQ
- Leopold, C. (2008). Plant 2 [CC-BY]. https://www.flickr.com/photos/97708873@N00/2669714872
- Hawk-Eye (2013). Schéma de la traduction d’un ARNm (ARN messager) en protéine [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Protein_synthesis-fr.svg
- LadyofHats (2012). Organization levels from cell until organism [CC0]. https://commons.wikimedia.org/wiki/File:Organization_levels_mouse.svg
- Ernst, R., T. Lautenschlager, M.F. Branquima et M. Holting (2020). At the edge of extinction: a first herpetological assessment of the proposed Serra do Pingano Rainforest National Park in Uige Province, northern Angola [CC-BY]. Zoosystematics and Evolution, 96(1), 237-262. https://doi.org/10.3897/zse.96.51997
- Leavitt, S., F. Fernández-Mendoza, S. Pérez-Ortega, P. Divakar, T. Lumbsch et L.S. Clair (2013). DNA barcode identification of lichen-forming fungal species in the Rhizoplaca melanophthalma species-complex (Lecanorales, Lecanoraceae), including five new species [CC-BY]. https://mycokeys.pensoft.net/article/1201/list/2/
- bayucca (2011). Amazon Poison Dartfrog (Ranitomeya variabilis) [CC-BY-NC-SA]. https://www.flickr.com/photos/dominikhofer/5519764529/in/photostream/
- Debivort (2006). Various coquina shells. Individuals in the mollusk species Donax variabilis show diverse coloration and patterning in their phenotypes [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Coquina_variation3.jpg
- Jeevan, J. (2014). Agriocnemis pieris Laidlaw, 1919 [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Agriocnemis_pieris_female_red_form_on_kadavoor.jpg
- Jeevan, J. (2010). White Dartlet (Agriocnemis pieris); commonly found in wet grasslands, especially among laterite outcrops [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Agriocnemis_pieris_female_at_kadavoor.jpg
- Aidannnuigalway (2015). The Kerry slug Geomalacus maculosus occurs in two colour morphs in Ireland [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Kerry_slugs_-_forest_and_bog_phenotypes.jpg
- University of California Museum of Paleontology. Understanding Evolution [CC-BY-NC-SA]. https://evolution.berkeley.edu/teach-resources/genes-environment-phenotype/
- Mahmoud, M., N. Gobet, D.I. Cruz-Dávalos, N. Mounier, C. Dessimoz et F.J. Sedlazeck (2019). Structural variant calling: the long and the short of it. Genome Biology, 20(1). https://doi.org/10.1186/s13059-019-1828-7
- Clark, M.A., M. Douglas et J. Choi (2018). Mutations can lead to changes in the protein sequence encoded by the DNA [CC-BY]. https://openstax.org/books/biology-2e/pages/14-6-dna-repair
- National Human Genome Research Institute (2007). Drawing showing what happens in gene duplication [domaine public]. https://commons.wikimedia.org/wiki/File:Gene-duplication.png
- Genome Research Limited (2021). Illustration to show the inheritance of dominant and recessive alleles for eye colour [CC-BY]. https://www.yourgenome.org/facts/what-are-dominant-and-recessive-alleles/
- mnakano (2015). Laws of Inheritance Ch 8.2 Exercises [CC-BY]. https://opentextbc.ca/biology/chapter/8-2-laws-of-inheritance/
- ACC-BioinnovationLab (2015). Diagram showing a part of the normal and mutated DNA strand, resulting change in mRNA and amino acid sequence [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Point-Mutation-Sickle-Cell-Normal_and_Mutated-Hemoglobin.png
- Grib, D. (2015). III [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Risk-Factors-for-Sickle-Cell-Anemia_(1)2.jpg
- Khan Academy. Polygenic inheritance [CC-BY-NC-SA]. https://www.khanacademy.org/science/biology/classical-genetics/variations-on-mendelian-genetics/a/polygenic-inheritance-and-environmental-effects
- B. Neuffer, M. Schorsch, S. Hameister, J. Knuesting, J. Selinski et R. Scheibe (2020). Habitus of individual plants cultivated under different light conditions [CC-BY-NC]. https://peerj.com/articles/9226/
- Harrison, A. (2008). The Aviary at Cooper Park [CC-BY-SA]. https://www.geograph.org.uk/photo/813390
- Amgueddfa Cymru (2008). Garden Tiger Moth [CC-BY-NC-SA]. https://www.flickr.com/photos/museumwales/2384239799
- Gould, J. Darwin’s finches or Galapagos finches. Darwin, 1845. Journal of researches into the natural history and geology of the countries visited during the voyage of H.M.S. Beagle round the world, under the Command of Capt. Fitz Roy [domaine public]. https://commons.wikimedia.org/wiki/File:Darwin%27s_finches.jpeg
- Cerlin Ng (2015). Cordia lutea [CC-BY-NC-SA]. https://www.flickr.com/photos/89906643@N06/21157839973/in/photolist-etFjqw-2dQvLJ4-GGeiRW-GGeiPw-iuHX4B-iuH2Dt-7uDBYj-6e85QV-AALxrD-Asdy8b-w2YxhG-BvELSP-ArVUx8-z9dBJA-ApK5u7-yeDsGn-wiVFu5-w2YxDJ-Bvxn4S-wk34tM-w36pUv-bWwjoc-2nwU1c9-2nwZgoT-8ovUU9-fAau7R-fApQQ3-fApPC1-p3MwWj-p3MGwb/
- Veronesi, F. (2014). Black-bellied Seedcracker – Kakum – Ghana_S4E2847 [CC-BY-SA]. https://commons.wikimedia.org/wiki/File:Black-bellied_Seedcracker_-_Kakum_-_Ghana_S4E2847_(22799387200).jpg
- vonHoldt, B.M., R.Y. Kartzinel, C.D. Huber, V. Le Underwood, Y. Zhen, K. Ruegg, K.E. Lohmueller et T.B. Smith (2018). Growth factor gene IGF1 is associated with bill size in the black-bellied seedcracker Pyrenestes ostrinus [CC-BY]. Nature Communications, 9, 12. https://doi.org/10.1038/s41467-018-07374-9
- Todesco, M., N. Bercovich, A. Kim, I. Imerovski, G.L. Owens, O.D. Ruiz, S.V. Holalu, L.L. Madilao, M. Jahani, J.S. Legare, B.K. Blackman et L.H. Rieseberg (2022). Genetic basis and dual adaptive role of floral pigmentation in sunflowers [CC-BY]. Elife, 11, 26. https://doi.org/10.7554/eLife.72072
- Bogomolov.PL (2005). Populus euphratica (syn. P. diversifolia), Ekhiin-Gol oazis, Shinejinst sum, Bayankhongor province, Mongolia, Gobi desert [domaine public]. https://commons.wikimedia.org/wiki/File:Populus_diversifolia,_Ekhiin-Gol_oazis,_Shinejinst_sum,_Bayankhongor_province,_Mongolia,_Gobi_desert.JPG
- Ma, T., J. Wang, G. Zhou, Z. Yue, Q. Hu, Y. Chen, B. Liu, Q. Qiu, Z. Wang, J. Zhang, K. Wang, D. Jiang, C. Gou, L. Yu, D. Zhan, R. Zhou, W. Luo, H. Ma, Y. Yang, S. Pan, D. Fang, Y. Luo, X. Wang, G. Wang, J. Wang, Q. Wang, X. Lu, Z. Chen, J. Liu, Y. Lu, Y. Yin, H. Yang, R.J. Abbott, Y. Wu, D. Wan, J. Li, T. Yin, M. Lascoux, S.P. Difazio, G.A. Tuskan, J. Wang et J. Liu (2013). Genomic insights into salt adaptation in a desert poplar [CC-BY]. Nature Communications, 4(1). https://doi.org/10.1038/ncomms3797
- Thomas, K. (2007). A Dark-eyed Junco subspecies – the Slate-colored Junco (Junco hyemalis hyemalis) [domaine public]. https://commons.wikimedia.org/wiki/File:Dark-eyed_Junco-27527.jpg
- Stager, M., N.R. Senner, D.L. Swanson, M.D. Carling, D.K. Eddy, T.J. Greives et Z.A. Cheviron (2021). Temperature heterogeneity correlates with intraspecific variation in physiological flexibility in a small endotherm [CC-BY]. Nature Communications, 12(1). https://doi.org/10.1038/s41467-021-24588-6

